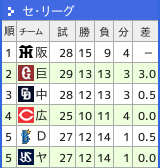
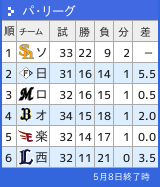
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
library(Design)
result <- lrm(y ~ x1 + x2, dat)
summary(result)ではエラーで
result としなくてはいけない
Nagelkerke's R二乗、tau, gamma, 回帰係数、他色々出る。
glm関数と同じだが…
result$deviance で尤度、result$linear.predictorsで予測子など。
str(result) を見るといろいろ調べられるようだが、今日はこの辺にしてやろう
result <- lrm(y ~ x1 + x2, dat)
summary(result)ではエラーで
result としなくてはいけない
Nagelkerke's R二乗、tau, gamma, 回帰係数、他色々出る。
glm関数と同じだが…
result$deviance で尤度、result$linear.predictorsで予測子など。
str(result) を見るといろいろ調べられるようだが、今日はこの辺にしてやろう
PR
# 分析実行
result <- glm(y~x1+x2, dat, family=binomial)
# 予測子を取り出してx軸のデータにする
xaxis1 <- result$linear.predictor
## yaxis1 <- result$fitted.values #予測値をとりだす (別にここではいらない)
# 曲線描画用のx軸データを生成する。lengthを多く指定すればよりなめらか
xaxis2 <- seq(from=min(xaxis1), to=max(xaxis1), length=200)
# 曲線用の予測値データを生成する
yaxis2 <- exp(xaxis2)/(1+exp(xaxis2)) #生成データから予測値を出す。分子に注意
# プロット
plot(xaxis1, dat$y) #元の予測子と実測データをプロット
lines(xaxis2, yaxis2) #生成データで曲線を描画する
# 少しきれいにしたプロット
pdf("LoGraph.pdf", family = "Japan1GothicBBB") #pdfファイルの指定と日本語フォント指定
plot(xaxis1, dat$y, pch = ifelse(dat$y=="1", 16, 1), xlab = "独立変数", ylab = "従属変数") #pch=...で1は黒丸、0は白丸に指定。xlabでx軸の名前、ylabでy軸の名前を指定
lines(xaxis2, yaxis2) #生成データで曲線を描画する
dev.off() #作図デバイスの終了
# pdfで出力。
グラフ
# jpegで出力。pdfの方が軽くてきれい

なお、元データはRによる統計解析p.181より
result <- glm(y~x1+x2, dat, family=binomial)
# 予測子を取り出してx軸のデータにする
xaxis1 <- result$linear.predictor
## yaxis1 <- result$fitted.values #予測値をとりだす (別にここではいらない)
# 曲線描画用のx軸データを生成する。lengthを多く指定すればよりなめらか
xaxis2 <- seq(from=min(xaxis1), to=max(xaxis1), length=200)
# 曲線用の予測値データを生成する
yaxis2 <- exp(xaxis2)/(1+exp(xaxis2)) #生成データから予測値を出す。分子に注意
# プロット
plot(xaxis1, dat$y) #元の予測子と実測データをプロット
lines(xaxis2, yaxis2) #生成データで曲線を描画する
# 少しきれいにしたプロット
pdf("LoGraph.pdf", family = "Japan1GothicBBB") #pdfファイルの指定と日本語フォント指定
plot(xaxis1, dat$y, pch = ifelse(dat$y=="1", 16, 1), xlab = "独立変数", ylab = "従属変数") #pch=...で1は黒丸、0は白丸に指定。xlabでx軸の名前、ylabでy軸の名前を指定
lines(xaxis2, yaxis2) #生成データで曲線を描画する
dev.off() #作図デバイスの終了
# pdfで出力。
グラフ
# jpegで出力。pdfの方が軽くてきれい
なお、元データはRによる統計解析p.181より
自分用に手順を書いておく
#1. まず何はともあれ実行しておく。因子数は理論に従う
result <- factanal(dat, factors=3, rotation="varimax", scores="regression")
#2. その上で、他の因子数モデルを実行し、カイ二乗値を比較する
result3 <- factanal(..factors=3,..)
result4 <- factanal(..factors=4,..)
...
## カイ二乗値が有意水準を超えたところが適切な因子数
#3. 適切な因子数で分析し、結果を見る
print(result, cut=0.001)
## cut=0.001とすることで、負荷量で0.1未満の数値も表示される
共通性
1-result%uniqueness
## 1から独自性を引く
寄与率
resultの中の
Proportion Var
Cumulative Var # 累積寄与率
因子負荷量
print(sort.loadings(result), cut=0.001)
## 青木先生の並べ替え関数を使わせていただく。感謝
## プロマックス回転時の因子間相関
fcor <- factanal(dat,factors=3,rotation="none")
pro <- promax(loadings(fcor),m=3)
solve(t(pro$rotmat)%*%pro$rotmat)
#1. まず何はともあれ実行しておく。因子数は理論に従う
result <- factanal(dat, factors=3, rotation="varimax", scores="regression")
#2. その上で、他の因子数モデルを実行し、カイ二乗値を比較する
result3 <- factanal(..factors=3,..)
result4 <- factanal(..factors=4,..)
...
## カイ二乗値が有意水準を超えたところが適切な因子数
#3. 適切な因子数で分析し、結果を見る
print(result, cut=0.001)
## cut=0.001とすることで、負荷量で0.1未満の数値も表示される
共通性
1-result%uniqueness
## 1から独自性を引く
寄与率
resultの中の
Proportion Var
Cumulative Var # 累積寄与率
因子負荷量
print(sort.loadings(result), cut=0.001)
## 青木先生の並べ替え関数を使わせていただく。感謝
## プロマックス回転時の因子間相関
fcor <- factanal(dat,factors=3,rotation="none")
pro <- promax(loadings(fcor),m=3)
solve(t(pro$rotmat)%*%pro$rotmat)
1要因反復測定の分散分析をaov, nlmeパッケージのlme関数、lme4パッケージのlmer関数でやってみた。
lmeは線型混合モデル、lmerは混合効果モデルでの分析
dat <- read.table("http://personality-project.org/r/datasets/R.appendix3.data", header=T)
colnames(dat) <- c("no", "sbj", "x", "y") # 列名を短くする
aggregate(dat$y, list(dat$x), mean, na.rm=TRUE) # 平均
aggregate(dat$y, list(dat$x), sd, na.rm=TRUE) # 標準偏差
length(levels(dat$x)) # 水準数を調べる
levels(dat$x) # 水準の中身を調べる
# aovでやってみた
aovres <- aov(y ~ x + Error(sbj/x), dat)
## aov(y ~ x + sbj + Error(x:sbj), dat) # これでも同じ。
summary(aovres)
# lmeでやってみた
require(nlme)
lmeres <- lme(y ~ x, random = ~1|sbj/x, data=dat)
summary(lmeres)
anova(lmeres)
# lmerでやってみた
require(lme4)
lmerres <- lmer(y ~ x + (1|sbj), data=dat)
summary(lmerres)
anova(lmerres)
## lmerres2 <- lmer(y ~ x + (x|sbj), data=dat) # ランダム切片/傾きモデル
# 多重比較
#holm法
pairwise.t.test(dat$y, dat$x, p.adjust.method="holm", paired=TRUE)
# Tukey法。対応ありのデータでTukey法を使うのはだめ、と誰かが言ってた気がするが
require(multcomp)
summary(glht(aovres,linfct=mcp(x="Tukey"))) # aov関数の結果は受け取れず、エラーを返す
summary(glht(lmeres,linfct=mcp(x="Tukey")))
summary(glht(lmerres,linfct=mcp(x="Tukey")))
参考: http://gribblelab.org/2009/03/09/repeated-measures-anova-using-r/
愚痴: 分散分析というのはマイナーな手法なのか、解説がない。aovを使ったもの (タイプ1平方和) は多いが、タイプ3では計算できない。carパッケージのAnova関数を使えばタイプ3で実行できるが、今度は誤差項を指定できない。混合デザインに使えなくもないが。事後検定についても書いてあるところはないし、aov関数のように汎用性が高くないし、分析結果に変量効果 (random effect) が入らない (たぶん計算はしてる) 。
ほとんどのページがしれっとaovでのやり方を書いてるけどそれありなの?
lmeは線型混合モデル、lmerは混合効果モデルでの分析
dat <- read.table("http://personality-project.org/r/datasets/R.appendix3.data", header=T)
colnames(dat) <- c("no", "sbj", "x", "y") # 列名を短くする
aggregate(dat$y, list(dat$x), mean, na.rm=TRUE) # 平均
aggregate(dat$y, list(dat$x), sd, na.rm=TRUE) # 標準偏差
length(levels(dat$x)) # 水準数を調べる
levels(dat$x) # 水準の中身を調べる
# aovでやってみた
aovres <- aov(y ~ x + Error(sbj/x), dat)
## aov(y ~ x + sbj + Error(x:sbj), dat) # これでも同じ。
summary(aovres)
# lmeでやってみた
require(nlme)
lmeres <- lme(y ~ x, random = ~1|sbj/x, data=dat)
summary(lmeres)
anova(lmeres)
# lmerでやってみた
require(lme4)
lmerres <- lmer(y ~ x + (1|sbj), data=dat)
summary(lmerres)
anova(lmerres)
## lmerres2 <- lmer(y ~ x + (x|sbj), data=dat) # ランダム切片/傾きモデル
# 多重比較
#holm法
pairwise.t.test(dat$y, dat$x, p.adjust.method="holm", paired=TRUE)
# Tukey法。対応ありのデータでTukey法を使うのはだめ、と誰かが言ってた気がするが
require(multcomp)
summary(glht(aovres,linfct=mcp(x="Tukey"))) # aov関数の結果は受け取れず、エラーを返す
summary(glht(lmeres,linfct=mcp(x="Tukey")))
summary(glht(lmerres,linfct=mcp(x="Tukey")))
参考: http://gribblelab.org/2009/03/09/repeated-measures-anova-using-r/
愚痴: 分散分析というのはマイナーな手法なのか、解説がない。aovを使ったもの (タイプ1平方和) は多いが、タイプ3では計算できない。carパッケージのAnova関数を使えばタイプ3で実行できるが、今度は誤差項を指定できない。混合デザインに使えなくもないが。事後検定についても書いてあるところはないし、aov関数のように汎用性が高くないし、分析結果に変量効果 (random effect) が入らない (たぶん計算はしてる) 。
ほとんどのページがしれっとaovでのやり方を書いてるけどそれありなの?
青木先生のfrequency関数が極めて有用。
LatexのデフォルトをFALSEにして新しく定義した。
以下、使用法の覚書
# とりあえず全部書き出すとき
layout(matrix(1:6, 3, 2, byrow=TRUE)) #3x2のレイアウト。事前にncol(データフレーム) で列数を調べよう
frq(1:5, iris)
# 複数のグラフを別々に得るときは
frq(1:5, iris, plot="A") #pdfファイルで作業ディレクトリに保存される
# hist関数の階級幅を利用して度数分布表を書く
x <- rnorm(1000, mean=100, sd=20)
hp <- hist(x, right=F)
b1 <- hp$breaks
a <- b1[-length(b1)]
b <- b1[-1]
b2 <- paste(a, b, sep="-")
names(hp$counts) <- b2
# 度数分布表
data.frame(hp$counts)
# barplotで描く
barplot(hp$counts)
LatexのデフォルトをFALSEにして新しく定義した。
以下、使用法の覚書
# とりあえず全部書き出すとき
layout(matrix(1:6, 3, 2, byrow=TRUE)) #3x2のレイアウト。事前にncol(データフレーム) で列数を調べよう
frq(1:5, iris)
# 複数のグラフを別々に得るときは
frq(1:5, iris, plot="A") #pdfファイルで作業ディレクトリに保存される
# hist関数の階級幅を利用して度数分布表を書く
x <- rnorm(1000, mean=100, sd=20)
hp <- hist(x, right=F)
b1 <- hp$breaks
a <- b1[-length(b1)]
b <- b1[-1]
b2 <- paste(a, b, sep="-")
names(hp$counts) <- b2
# 度数分布表
data.frame(hp$counts)
# barplotで描く
barplot(hp$counts)