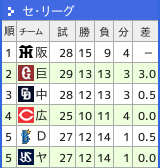
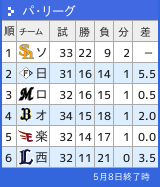
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
こっちとあわせて使おう
# split関数でデータフレームを分割
sp <- split(iris, iris$Species)
# summaryで内容がわかる
summary(sp)
# データフレームを3つつくる
iris.set <- sp$setosa
iris.ve <- sp$versicolor
iris.vi <- sp$virginica
# psychパッケージのmulti.hist関数でそれぞれの群のヒストグラムを描く
win.graph()
multi.hist(iris.set)
# 自分の定義したfrq関数では frq(1:5, iris.set)
win.graph() #別のウィンドウに描く
multi.hist(iris.ve)
win.graph() #別のウィンドウに描く
multi.hist(iris.vi)
multi.hist関数の引数もメモしておこう
(あまり汎用性はない。こっちで作ってもいいかも…)
### subset関数で分割してもよし
# 群の名前を調べておく。ついでに群の数も調べておく
grpname <- levels(iris$Species)
length(levels(iris$Species))
#subset関数で群を分けたデータフレームをつくる
iris.se <- subset(iris, Species == grpname[1]).
# split関数でデータフレームを分割
sp <- split(iris, iris$Species)
# summaryで内容がわかる
summary(sp)
# データフレームを3つつくる
iris.set <- sp$setosa
iris.ve <- sp$versicolor
iris.vi <- sp$virginica
# psychパッケージのmulti.hist関数でそれぞれの群のヒストグラムを描く
win.graph()
multi.hist(iris.set)
# 自分の定義したfrq関数では frq(1:5, iris.set)
win.graph() #別のウィンドウに描く
multi.hist(iris.ve)
win.graph() #別のウィンドウに描く
multi.hist(iris.vi)
multi.hist関数の引数もメモしておこう
(あまり汎用性はない。こっちで作ってもいいかも…)
Usage
multi.hist(x,nrow=NULL, ncol=NULL,density=TRUE,main="Histogram, Density, and Normal Fit")
Arguments
x 行列かデータフレーム
nrow 作図デバイスの行数
ncol 作図デバイスの列数
density 正規分布曲線と分布
main グラフのタイトル
multi.hist(x,nrow=NULL, ncol=NULL,density=TRUE,main="Histogram, Density, and Normal Fit")
Arguments
x 行列かデータフレーム
nrow 作図デバイスの行数
ncol 作図デバイスの列数
density 正規分布曲線と分布
main グラフのタイトル
### subset関数で分割してもよし
# 群の名前を調べておく。ついでに群の数も調べておく
grpname <- levels(iris$Species)
length(levels(iris$Species))
#subset関数で群を分けたデータフレームをつくる
iris.se <- subset(iris, Species == grpname[1]).
PR
# クロス集計表をしこたまつくる
for(i in 2:50){
cat("\n",colnames(dat[i])," x ", colnames(dat[1]),"\n") # 集計に使った変数名を出力
print(xtabs(~ dat[,i] + dat[,1], dat))
}
# 合計欄つきでしこたまつくる
for(i in 2:50){
cat("\n",colnames(dat[i])," x ", colnames(dat[1]),"\n")
print(addmargins(xtabs(~ dat[,i] + dat[,1], dat)))
}
# 列パーセントのクロス集計をしこたまつくる
for(i in 2:50){
cat("\n",colnames(dat[i])," x ", colnames(dat[1]),"\n")
xn <- addmargins(xtabs(~ dat[,i] + dat[,1], dat))
print(round(t(t(xn)/xn[nrow(xn),]*100),2))
}
# 行パーセントの以下略
for(i in 2:50){
cat("\n",colnames(dat[i])," x ", colnames(dat[1]),"\n")
xn <- addmargins(xtabs(~ dat[,i] + dat[,1], dat))
print(round((xn/xn[, ncol(xn)]*100),2))
}
# クロス集計、度数、パーセント、カイ二乗検定、Fisherの正確確率検定。
# cat関数で適当にセパレータ ("===")を入れる。改行は\n
## Fisherのはメモリが足りなくてできないことがある
for(i in 2:50){
cat("\n\n========================\n")
cat(colnames(dat[i])," x ", colnames(dat[1]),"\n")
x <- xtabs(~ dat[,i]+ dat[,1], dat)
cat("\n===\n")
print(ftable(addmargins(x)))
xn <- ftable(addmargins(x))
cat("\n===\n")
print(round(t(t(xn)/xn[nrow(xn),]*100),2))
cat("\n===\n")
print(round((xn/xn[, ncol(xn)]*100),2))
(crr <- chisq.test(x))
cat("\n\nCHISQ\n")
(cr <- my.chisq.test(x))
print(cr)
cat("\n===\n")
cat("EXPECTED\n")
print(crr$expected)
cat("\n===\n")
summary(cr)
cat("\n\nMYFISHER\n")
fx <- my.fisher(x)
cat("\nMYFISHER$Pearson")
print(fx$p.Pearson)
cat("\nMYFISHER$Fisher")
print(fx$p.Fisher)
cat("\n========================\n")
}
青木先生のfrequency関数が極めて有用。
LatexのデフォルトをFALSEにして新しく定義した。
以下、使用法の覚書
# とりあえず全部書き出すとき
layout(matrix(1:6, 3, 2, byrow=TRUE)) #3x2のレイアウト。事前にncol(データフレーム) で列数を調べよう
frq(1:5, iris)
# 複数のグラフを別々に得るときは
frq(1:5, iris, plot="A") #pdfファイルで作業ディレクトリに保存される
# hist関数の階級幅を利用して度数分布表を書く
x <- rnorm(1000, mean=100, sd=20)
hp <- hist(x, right=F)
b1 <- hp$breaks
a <- b1[-length(b1)]
b <- b1[-1]
b2 <- paste(a, b, sep="-")
names(hp$counts) <- b2
# 度数分布表
data.frame(hp$counts)
# barplotで描く
barplot(hp$counts)
LatexのデフォルトをFALSEにして新しく定義した。
以下、使用法の覚書
# とりあえず全部書き出すとき
layout(matrix(1:6, 3, 2, byrow=TRUE)) #3x2のレイアウト。事前にncol(データフレーム) で列数を調べよう
frq(1:5, iris)
# 複数のグラフを別々に得るときは
frq(1:5, iris, plot="A") #pdfファイルで作業ディレクトリに保存される
# hist関数の階級幅を利用して度数分布表を書く
x <- rnorm(1000, mean=100, sd=20)
hp <- hist(x, right=F)
b1 <- hp$breaks
a <- b1[-length(b1)]
b <- b1[-1]
b2 <- paste(a, b, sep="-")
names(hp$counts) <- b2
# 度数分布表
data.frame(hp$counts)
# barplotで描く
barplot(hp$counts)
標準誤差の関数を見つけたのでメモしておく
# 関数
se <- function(x)
{
y <- x[!is.na(x)] # remove the missing values
sqrt(var(as.vector(y))/length(y))
}
# sapplyにした
sapply(dat, function(x) sqrt(var(as.vector(na.omit(x)))/length(na.omit(x))))
こっちも変えた
http://scratchhit.pazru.com/Entry/20/
# 関数
se <- function(x)
{
y <- x[!is.na(x)] # remove the missing values
sqrt(var(as.vector(y))/length(y))
}
# sapplyにした
sapply(dat, function(x) sqrt(var(as.vector(na.omit(x)))/length(na.omit(x))))
こっちも変えた
http://scratchhit.pazru.com/Entry/20/
cor関数で行列、2変数を指定してcor.testをすると無相関検定ができる
psychパッケージのcorr.testで複数の相関行列、サンプルサイズ、P値を算出してくれる…が、小数点第2位までしか出してくれない
どういう仕組みかわからないが (知ってる人は知ってるんだろうけど)
res <- corr.test # resにcorr.testの結果を入れて
res$r # とすると小数点以下もたくさん出力できる (options(digits=6) も効く) 。
# pはres$p, nはres$n, t統計量もres$pでわかる
青木先生のmycor関数が有用。感謝
method="pearson", "kendall", "spearman"が選択可能
#irisの1列目1-5行にNAを入れる
irisna <- iris
irisna[1:5,1] <- NA
# cor関数はfactor変数が入っているとエラーを返す
# リストワイズ除去した相関。NAを含むケースを全て除く
cor(irisna[1:4], use="c")
mycor(1:4, irisna, latex=FALSE)
nrow(na.omit(irisna[1:4])) # サンプルサイズ
# ペアワイズ除去。ペアごとにNAを除く
cor(irisna[1:4], use="p")
mycor(1:4, irisna, use="pairwise", latex=FALSE)
# グループ別に相関を求める
by(irisna[1:4], iris[,5], cor, use="c") # use="p"でペアワイズ
psychパッケージのcorr.testで複数の相関行列、サンプルサイズ、P値を算出してくれる…が、
どういう仕組みかわからないが (知ってる人は知ってるんだろうけど)
res <- corr.test # resにcorr.testの結果を入れて
res$r # とすると小数点以下もたくさん出力できる (options(digits=6) も効く) 。
# pはres$p, nはres$n, t統計量もres$pでわかる
青木先生のmycor関数が有用。感謝
method="pearson", "kendall", "spearman"が選択可能
#irisの1列目1-5行にNAを入れる
irisna <- iris
irisna[1:5,1] <- NA
# cor関数はfactor変数が入っているとエラーを返す
# リストワイズ除去した相関。NAを含むケースを全て除く
cor(irisna[1:4], use="c")
mycor(1:4, irisna, latex=FALSE)
nrow(na.omit(irisna[1:4])) # サンプルサイズ
# ペアワイズ除去。ペアごとにNAを除く
cor(irisna[1:4], use="p")
mycor(1:4, irisna, use="pairwise", latex=FALSE)
# グループ別に相関を求める
by(irisna[1:4], iris[,5], cor, use="c") # use="p"でペアワイズ