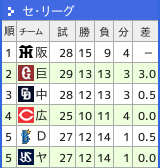
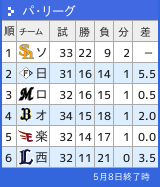
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
# 足立浩平 (2006). 多変量データ解析法――心理・教育・社会系のための入門―― ナカニシヤ出版の例より
# 因子分析と主成分分析は似て非なる方法であることに注意
# 以下のサイトを参考。というかRコマンダーでの例をそのままコードにしただけ。感謝
http://www.nanzan-u.ac.jp/~kamiya/r/content/rcmdrprin.html
http://www.statmethods.net/advstats/factor.html
# 表 3.1 p24
ppt <- c(88,52,77,35,60,97,48,66,78)
wrt <- c(70,78,87,40,43,95,62,66,50)
int <- c(65,88,89,43,40,91,83,65,48)
dat <- data.frame(ppt, wrt, int)
mean(dat)
sapply(dat, var)
fit <- princomp(~ppt+wrt+int, cor=F, data=dat) # 相関行列をもとにした分析をするにはcor=Tとする
fit2 <- prcomp(dat, cor=F)
# 主成分得点 表3.1 p22
# 単にprincomp(dat) でも同じ
fit$scores
fit2$x
# 各主成分の係数 表3.5 p32
loadings(fit)
unclass(loadings(fit)) # 行列にしただけ
fit2$rotation
# 固有ベクトルなので以下と同じ
eigen(cov(dat))$vectors
# 各主成分得点どうしの共分散 表3.6 p32
cov(fit$scores)
round(cov(fit$scores),1) # みやすく四捨五入
round(cov(fit2$x),1)
# 成分行列 (いわゆる負荷量)
t(fit$sd * t(fit$loadings ) )[, drop = FALSE]
t(fit2$sdev * t(fit2$rotation ) )[, drop = FALSE]
# 結果が異なる。おそらくprcompは Unlike princomp, variances are computed with the usual divisor N - 1. であるため、標準偏差の値が異なる
fit$sd
fit2$sdev
sqrt(fit2$sdev^2 *(8/9)) # 不偏分散を標本に戻すと一致
# 説明率、累積説明率 図3.6 p27
summary(fit)
print(summary(fit2), digits=10) #
基本的には同じ結果だが、prcompのsummaryは小数点第5桁までで区切っているのがわかる
# 固有値
fit$sd^2
fit2$sdev^2
# 固有値なので以下と同じ
eigen(cov(dat))$value
## おまけ。psychパッケージでもやってみる
fit2 <- princomp(~ppt+wrt+int, cor=T, data=dat) # 相関行列を使うよう指定してprincomp関数で実行
library(psych)
fit3 <- principal(dat, nfactors=3, rotate=F, scores=T)# psychパッケージでは標準化した相関行列がデフォルトで用いられる。回転もデフォルトだとバリマックス回転が行われる
summary(fit2) # princomp関数
print(fit3, digits=5) # psychパッケージのprincipal関数
fit2$scores
fit3$scores # psychパッケージでは標準化した主成分得点が算出される
## 覚書
prcomp関数は各個体の平均から偏差を求めるQモード法。対してprincomp, principal関数はRモード法。Qモード法は変数の数 > サンプルサイズ でも実行できる。
# 因子分析と主成分分析は似て非なる方法であることに注意
# 以下のサイトを参考。というかRコマンダーでの例をそのままコードにしただけ。感謝
http://www.nanzan-u.ac.jp/~kamiya/r/content/rcmdrprin.html
http://www.statmethods.net/advstats/factor.html
# 表 3.1 p24
ppt <- c(88,52,77,35,60,97,48,66,78)
wrt <- c(70,78,87,40,43,95,62,66,50)
int <- c(65,88,89,43,40,91,83,65,48)
dat <- data.frame(ppt, wrt, int)
mean(dat)
sapply(dat, var)
fit <- princomp(~ppt+wrt+int, cor=F, data=dat) # 相関行列をもとにした分析をするにはcor=Tとする
fit2 <- prcomp(dat, cor=F)
# 主成分得点 表3.1 p22
# 単にprincomp(dat) でも同じ
fit$scores
fit2$x
# 各主成分の係数 表3.5 p32
loadings(fit)
unclass(loadings(fit)) # 行列にしただけ
fit2$rotation
# 固有ベクトルなので以下と同じ
eigen(cov(dat))$vectors
# 各主成分得点どうしの共分散 表3.6 p32
cov(fit$scores)
round(cov(fit$scores),1) # みやすく四捨五入
round(cov(fit2$x),1)
# 成分行列 (いわゆる負荷量)
t(fit$sd * t(fit$loadings ) )[, drop = FALSE]
t(fit2$sdev * t(fit2$rotation ) )[, drop = FALSE]
# 結果が異なる。おそらくprcompは Unlike princomp, variances are computed with the usual divisor N - 1. であるため、標準偏差の値が異なる
fit$sd
fit2$sdev
sqrt(fit2$sdev^2 *(8/9)) # 不偏分散を標本に戻すと一致
# 説明率、累積説明率 図3.6 p27
summary(fit)
print(summary(fit2), digits=10) #
基本的には同じ結果だが、prcompのsummaryは小数点第5桁までで区切っているのがわかる
# 固有値
fit$sd^2
fit2$sdev^2
# 固有値なので以下と同じ
eigen(cov(dat))$value
## おまけ。psychパッケージでもやってみる
fit2 <- princomp(~ppt+wrt+int, cor=T, data=dat) # 相関行列を使うよう指定してprincomp関数で実行
library(psych)
fit3 <- principal(dat, nfactors=3, rotate=F, scores=T)# psychパッケージでは標準化した相関行列がデフォルトで用いられる。回転もデフォルトだとバリマックス回転が行われる
summary(fit2) # princomp関数
print(fit3, digits=5) # psychパッケージのprincipal関数
fit2$scores
fit3$scores # psychパッケージでは標準化した主成分得点が算出される
## 覚書
prcomp関数は各個体の平均から偏差を求めるQモード法。対してprincomp, principal関数はRモード法。Qモード法は変数の数 > サンプルサイズ でも実行できる。
PR
Comment
Trackback
Trackback URL
Comment form