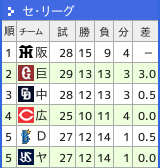
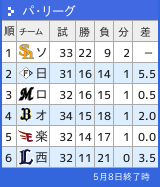
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
dat <- data.frame(
s=factor(c("s1", "s2", "s3", "s4", "s5", "s6", "s7")),
a=factor(c(rep("a1", 4), rep("a2", 3))),
b1c1=c(2,6,5,7,1,2,5), b1c2=c(5,7,9,9,2,1,4), b1c3=c(9,10,13,14,1,5,3),
b2c1=c(6,6,8,10,5,3,4), b2c2=c(3,6,5,8,5,6,6), b2c3=c(6,7,5,6,5,5,9)
)
options(contrasts = c("contr.sum", "contr.sum"))
lmres <- lm(cbind(b1c1, b1c2, b1c3, b2c1, b2c2, b2c3) ~ a, data = dat)
library(car)
bfact <- factor(rep(c("b1", "b2"), c(3, 3)))
cfact <- factor(rep(c("c1", "c2", "c3"), 2))
idata <- data.frame(bfact, cfact)
Anovares <- Anova(lmres, idata = idata , idesign = ~ bfact*cfact, type = 3)
summary(Anovares)
# aovと比較
library(reshape)
dat.aov <- melt(dat, id=c("s", "a"))
b <- c(rep(c("b1", "b2"), each=21))
c <- c(rep(c("c1", "c2", "c3"), each=7), rep(c("c1", "c2", "c3"), each=7))
dat.aov <- data.frame(dat.aov[c(1, 2, 4)], b, c)
names(dat.aov) <- c("s", "a", "result", "b", "c")
summary(aov(result ~ a * b * c + Error(s:a + b:s:a + c:s:a + b:c:s:a), dat.aov))
PR
carパッケージのAnova関数で混合要因の分散分析
データはデータ解析テクニカルブック
SPFp.qデザイン データ数が同じ(p.107)
dat <- data.frame(
s=factor(c("s1","s2","s3","s4","s5")),
a=factor(c(rep("a1",5), rep("a2",5))),
b1=c(3,3,1,3,5,3,5,2,4,6), b2=c(4,3,4,5,7,2,6,3,6,4), b3=c(6,6,6,4,8,3,2,3,6,5), b4=c(5,7,8,7,9,2,3,3,4,6)
)
dat
aggregate(dat[3:6], list(dat[,2]), sum)
# メインの分析
options(contrasts = c("contr.sum", "contr.sum"))
lmres <- lm(cbind(b1, b2, b3, b4) ~ a , contrasts = list(a = contr.sum), data = dat)
bfact <- as.factor(c("b1", "b2", "b3", "b4"))
idat <- data.frame(bfact)
library(car)
Anovares <- Anova(lmres, idata = idat, idesign = ~ bfact, type = 3)
summary(Anovares)
# aovと比較
library(reshape)
dat.aov <- melt(dat, id=c("s", "a"))
names(dat.aov) <- c("s", "a", "b", "result")
print(summary(aov(result ~ a * b + Error(s:a + s:a:b), dat.aov)))
# 作図のため、平均値を出して行列にする
agdat <- aggregate(dat[3:6], list(dat[,2]), mean)
mdat <- as.matrix(agdat[2:5])
# 折れ線で作図
matplot(mdat, type="b", pch=1:4, xaxt="n")
axis(1, at=1:2, labels=c("a1", "a2"))
legend("topright", legend=c("b1", "b2", "b3", "b4"), lty=1:4, pch=1:4, col=1:4)
# 棒グラフで作図
rownames(mdat) <- c("a1", "a2")
palette(grey.colors(nrow(t(mdat)))) # a要因を横軸にしたいのでtで転置
barplot(t(mdat), beside=T)
legend("topright", rownames(t(mdat)), fill = 1:4)
## 事後検定 テクニカルブックとは違って水準別
## b要因 (被験者内要因) の水準ごと
lmab1 <- lm(b1 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab1 <- Anova(lmab1, type = 3)
lmab2 <- lm(b2 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab2 <- Anova(lmab2, type = 3)
lmab3 <- lm(b3 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab3 <- Anova(lmab3, type = 3)
lmab4 <- lm(b4 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab4 <- Anova(lmab4, type = 3)
Anv.ab1
Anv.ab2
Anv.ab3
Anv.ab4
## 事後検定
## a要因 (被験者間要因) の水準ごと、aの1
levels(dat$a)
data1 <- subset(dat, a=="a1")
data1
lmba1 <- lm(cbind(b1, b2, b3, b4) ~ 1, contrasts=contr.sum, data = data1)
Anv.a1 <- Anova(lmba1, idata = idat, idesign = ~ bfact2, type = 3)
summary(Anv.a1)
## a要因 (被験者間要因) の水準ごと、aの2
data2 <- subset(dat, a=="a2")
data2
lmba2 <- lm(cbind(b1, b2, b3, b4) ~ 1, contrasts=contr.sum, data = data2)
Anv.a2 <- Anova(lmba2, idata = idat, idesign = ~ bfact2, type = 3)
summary(Anv.a2)
##多重比較 単純効果のあったa1で
#holm法
datpa <- stack(data1[3:6])
datpa$s <- rep(data1[,2], 4)
pttres <- pairwise.t.test(datpa$values, datpa$ind, p.adjust.method="holm", paired=TRUE)
pttres
#holm法2
p12 <- t.test(data1$b1, data1$b2, paired = T)
p13 <- t.test(data1$b1, data1$b3, paired = T)
p14 <- t.test(data1$b1, data1$b4, paired = T)
p23 <- t.test(data1$b2, data1$b3, paired = T)
p24 <- t.test(data1$b2, data1$b4, paired = T)
p34 <- t.test(data1$b3, data1$b4, paired = T)
p.adjust(c(p12$p.value,p13$p.value,p14$p.value,p23$p.value,p24$p.value,p34$p.value), method = "holm")
データはデータ解析テクニカルブック
SPFp.qデザイン データ数が同じ(p.107)
dat <- data.frame(
s=factor(c("s1","s2","s3","s4","s5")),
a=factor(c(rep("a1",5), rep("a2",5))),
b1=c(3,3,1,3,5,3,5,2,4,6), b2=c(4,3,4,5,7,2,6,3,6,4), b3=c(6,6,6,4,8,3,2,3,6,5), b4=c(5,7,8,7,9,2,3,3,4,6)
)
dat
aggregate(dat[3:6], list(dat[,2]), sum)
# メインの分析
options(contrasts = c("contr.sum", "contr.sum"))
lmres <- lm(cbind(b1, b2, b3, b4) ~ a , contrasts = list(a = contr.sum), data = dat)
bfact <- as.factor(c("b1", "b2", "b3", "b4"))
idat <- data.frame(bfact)
library(car)
Anovares <- Anova(lmres, idata = idat, idesign = ~ bfact, type = 3)
summary(Anovares)
# aovと比較
library(reshape)
dat.aov <- melt(dat, id=c("s", "a"))
names(dat.aov) <- c("s", "a", "b", "result")
print(summary(aov(result ~ a * b + Error(s:a + s:a:b), dat.aov)))
# 作図のため、平均値を出して行列にする
agdat <- aggregate(dat[3:6], list(dat[,2]), mean)
mdat <- as.matrix(agdat[2:5])
# 折れ線で作図
matplot(mdat, type="b", pch=1:4, xaxt="n")
axis(1, at=1:2, labels=c("a1", "a2"))
legend("topright", legend=c("b1", "b2", "b3", "b4"), lty=1:4, pch=1:4, col=1:4)
# 棒グラフで作図
rownames(mdat) <- c("a1", "a2")
palette(grey.colors(nrow(t(mdat)))) # a要因を横軸にしたいのでtで転置
barplot(t(mdat), beside=T)
legend("topright", rownames(t(mdat)), fill = 1:4)
## 事後検定 テクニカルブックとは違って水準別
## b要因 (被験者内要因) の水準ごと
lmab1 <- lm(b1 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab1 <- Anova(lmab1, type = 3)
lmab2 <- lm(b2 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab2 <- Anova(lmab2, type = 3)
lmab3 <- lm(b3 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab3 <- Anova(lmab3, type = 3)
lmab4 <- lm(b4 ~ a, contrasts = list(a = contr.sum), data = dat)
Anv.ab4 <- Anova(lmab4, type = 3)
Anv.ab1
Anv.ab2
Anv.ab3
Anv.ab4
## 事後検定
## a要因 (被験者間要因) の水準ごと、aの1
levels(dat$a)
data1 <- subset(dat, a=="a1")
data1
lmba1 <- lm(cbind(b1, b2, b3, b4) ~ 1, contrasts=contr.sum, data = data1)
Anv.a1 <- Anova(lmba1, idata = idat, idesign = ~ bfact2, type = 3)
summary(Anv.a1)
## a要因 (被験者間要因) の水準ごと、aの2
data2 <- subset(dat, a=="a2")
data2
lmba2 <- lm(cbind(b1, b2, b3, b4) ~ 1, contrasts=contr.sum, data = data2)
Anv.a2 <- Anova(lmba2, idata = idat, idesign = ~ bfact2, type = 3)
summary(Anv.a2)
##多重比較 単純効果のあったa1で
#holm法
datpa <- stack(data1[3:6])
datpa$s <- rep(data1[,2], 4)
pttres <- pairwise.t.test(datpa$values, datpa$ind, p.adjust.method="holm", paired=TRUE)
pttres
#holm法2
p12 <- t.test(data1$b1, data1$b2, paired = T)
p13 <- t.test(data1$b1, data1$b3, paired = T)
p14 <- t.test(data1$b1, data1$b4, paired = T)
p23 <- t.test(data1$b2, data1$b3, paired = T)
p24 <- t.test(data1$b2, data1$b4, paired = T)
p34 <- t.test(data1$b3, data1$b4, paired = T)
p.adjust(c(p12$p.value,p13$p.value,p14$p.value,p23$p.value,p24$p.value,p34$p.value), method = "holm")
reshape関数とstack関数を使うやり方がある
reshape関数はもともと入っている変数名を使える
stack関数は並べ替えるだけ
reshape関数
これはもともとaov用
spfp.q1 <- data.frame(
a = factor(c(rep(1,20), rep(2,20))),
b = factor(rep(c(rep(1,5), rep(2,5), rep(3,5), rep(4,5)),2)),
s = factor(c(rep(1:5, 4), rep(6:10, 4))),
result = c(3,3,1,3,5, 4,3,4,5,7, 6,6,6,4,8, 5,7,8,7,9,
3,5,2,4,6, 2,6,3,6,4, 3,2,3,6,5, 2,3,3,4,6))
comment(spfp.q1) <- "2way, mixed (1within, 1between), balanced"
## データ解析テクニカルブックより、松井先生のコード。感謝
# 横長にする
spfp.q1.2 <- reshape(spfp.q1, idvar = c("a","s"), timevar = "b", direction = "wide")
colnames(spfp.q1.2) <- c("a", "s", "b1", "b2", "b3", "b4")
# reshape関数で元の縦長にする
spfp.q1.3 <- reshape(spfp.q1.2, varying = list(3:6), idvar = "s", timevar = "b", direction = "long")
## varyingで縦に並べる変数 (反復測定変数) を指定する。idvarで被験者要因。timevar は反復測定変数の水準名の変数
spfp.q1.3 # "b"と"s"が入れ替わっているが気にしない
stack関数
stack関数を使うと反復測定要因を縦に並べたデータフレームを簡単に作れる
personality-projectの説明がわかりやすい
recall.data <- read.table("http://personality-project.org/r/datasets/recall1.data",header=TRUE)
sums <- recall.data[,9:12] #縦に並べる列をとりだす。
sums # データチェック
stackeds <- stack(sums) #stack関数で並べかえる
stackeds #データチェック
後は変数名を入れてるだけ
numcases <- 27 #被験者数
numvariables <- 4 #反復要因の水準数
## 後は変数、水準名を整理しているだけ
recall.df=data.frame(recall=stackeds,
subj=factor(rep(paste("subj", 1:numcases, sep=""), numvariables)),
time=factor(rep(rep(c("short", "long"), c(numcases, numcases)), 2)),
study=factor(rep(c("d45", "d90"), c(numcases*2, numcases*2))))
reshape関数はもともと入っている変数名を使える
stack関数は並べ替えるだけ
reshape関数
これはもともとaov用
spfp.q1 <- data.frame(
a = factor(c(rep(1,20), rep(2,20))),
b = factor(rep(c(rep(1,5), rep(2,5), rep(3,5), rep(4,5)),2)),
s = factor(c(rep(1:5, 4), rep(6:10, 4))),
result = c(3,3,1,3,5, 4,3,4,5,7, 6,6,6,4,8, 5,7,8,7,9,
3,5,2,4,6, 2,6,3,6,4, 3,2,3,6,5, 2,3,3,4,6))
comment(spfp.q1) <- "2way, mixed (1within, 1between), balanced"
## データ解析テクニカルブックより、松井先生のコード。感謝
# 横長にする
spfp.q1.2 <- reshape(spfp.q1, idvar = c("a","s"), timevar = "b", direction = "wide")
colnames(spfp.q1.2) <- c("a", "s", "b1", "b2", "b3", "b4")
# reshape関数で元の縦長にする
spfp.q1.3 <- reshape(spfp.q1.2, varying = list(3:6), idvar = "s", timevar = "b", direction = "long")
## varyingで縦に並べる変数 (反復測定変数) を指定する。idvarで被験者要因。timevar は反復測定変数の水準名の変数
spfp.q1.3 # "b"と"s"が入れ替わっているが気にしない
stack関数
stack関数を使うと反復測定要因を縦に並べたデータフレームを簡単に作れる
personality-projectの説明がわかりやすい
recall.data <- read.table("http://personality-project.org/r/datasets/recall1.data",header=TRUE)
sums <- recall.data[,9:12] #縦に並べる列をとりだす。
sums # データチェック
stackeds <- stack(sums) #stack関数で並べかえる
stackeds #データチェック
後は変数名を入れてるだけ
numcases <- 27 #被験者数
numvariables <- 4 #反復要因の水準数
## 後は変数、水準名を整理しているだけ
recall.df=data.frame(recall=stackeds,
subj=factor(rep(paste("subj", 1:numcases, sep=""), numvariables)),
time=factor(rep(rep(c("short", "long"), c(numcases, numcases)), 2)),
study=factor(rep(c("d45", "d90"), c(numcases*2, numcases*2))))
どうもlinear.hypothesisとかいう関数を使うらしい
http://tolstoy.newcastle.edu.au/R/e2/help/07/09/26498.html
http://tolstoy.newcastle.edu.au/R/e2/help/07/09/26518.html)
検証は面倒なので、重要そうなところだけメモしておこう
linear.hypothesis(diversitylm.ok, c("(Intercept)=CategoryM"), idata=idata.df, idesign=~Season, iterms="Season")
diversitylm.okはlm関数の結果
diversitylm.ok <- lm(cbind(dLluvias, dNortes, dSecas) ~ Category, data=datalm.df)
第2引数で被験者間要因のどの水準で検定するか
idata=idata.dfは被験者内要因の水準名を格納している
idata.df <- data.frame(Season)
idesign...被験者内要因の数式
Season <- factor(c("Lluvias","Nortes","Secas"), levels=c("Lluvias","Nortes","Secas"))
iterms...は被験者内要因の要因名
結果は対比や多変量統計量が出るようだ
ボンフェローニの調整が必要だから、多変量多重比較しろなどと書いてある
結果は対比や多変量統計量が出るようだ
ボンフェローニの調整が必要だから、多変量多重比較しろなどと書いてある
1要因反復測定の分散分析をaov, nlmeパッケージのlme関数、lme4パッケージのlmer関数でやってみた。
lmeは線型混合モデル、lmerは混合効果モデルでの分析
dat <- read.table("http://personality-project.org/r/datasets/R.appendix3.data", header=T)
colnames(dat) <- c("no", "sbj", "x", "y") # 列名を短くする
aggregate(dat$y, list(dat$x), mean, na.rm=TRUE) # 平均
aggregate(dat$y, list(dat$x), sd, na.rm=TRUE) # 標準偏差
length(levels(dat$x)) # 水準数を調べる
levels(dat$x) # 水準の中身を調べる
# aovでやってみた
aovres <- aov(y ~ x + Error(sbj/x), dat)
## aov(y ~ x + sbj + Error(x:sbj), dat) # これでも同じ。
summary(aovres)
# lmeでやってみた
require(nlme)
lmeres <- lme(y ~ x, random = ~1|sbj/x, data=dat)
summary(lmeres)
anova(lmeres)
# lmerでやってみた
require(lme4)
lmerres <- lmer(y ~ x + (1|sbj), data=dat)
summary(lmerres)
anova(lmerres)
## lmerres2 <- lmer(y ~ x + (x|sbj), data=dat) # ランダム切片/傾きモデル
# 多重比較
#holm法
pairwise.t.test(dat$y, dat$x, p.adjust.method="holm", paired=TRUE)
# Tukey法。対応ありのデータでTukey法を使うのはだめ、と誰かが言ってた気がするが
require(multcomp)
summary(glht(aovres,linfct=mcp(x="Tukey"))) # aov関数の結果は受け取れず、エラーを返す
summary(glht(lmeres,linfct=mcp(x="Tukey")))
summary(glht(lmerres,linfct=mcp(x="Tukey")))
参考: http://gribblelab.org/2009/03/09/repeated-measures-anova-using-r/
愚痴: 分散分析というのはマイナーな手法なのか、解説がない。aovを使ったもの (タイプ1平方和) は多いが、タイプ3では計算できない。carパッケージのAnova関数を使えばタイプ3で実行できるが、今度は誤差項を指定できない。混合デザインに使えなくもないが。事後検定についても書いてあるところはないし、aov関数のように汎用性が高くないし、分析結果に変量効果 (random effect) が入らない (たぶん計算はしてる) 。
ほとんどのページがしれっとaovでのやり方を書いてるけどそれありなの?
lmeは線型混合モデル、lmerは混合効果モデルでの分析
dat <- read.table("http://personality-project.org/r/datasets/R.appendix3.data", header=T)
colnames(dat) <- c("no", "sbj", "x", "y") # 列名を短くする
aggregate(dat$y, list(dat$x), mean, na.rm=TRUE) # 平均
aggregate(dat$y, list(dat$x), sd, na.rm=TRUE) # 標準偏差
length(levels(dat$x)) # 水準数を調べる
levels(dat$x) # 水準の中身を調べる
# aovでやってみた
aovres <- aov(y ~ x + Error(sbj/x), dat)
## aov(y ~ x + sbj + Error(x:sbj), dat) # これでも同じ。
summary(aovres)
# lmeでやってみた
require(nlme)
lmeres <- lme(y ~ x, random = ~1|sbj/x, data=dat)
summary(lmeres)
anova(lmeres)
# lmerでやってみた
require(lme4)
lmerres <- lmer(y ~ x + (1|sbj), data=dat)
summary(lmerres)
anova(lmerres)
## lmerres2 <- lmer(y ~ x + (x|sbj), data=dat) # ランダム切片/傾きモデル
# 多重比較
#holm法
pairwise.t.test(dat$y, dat$x, p.adjust.method="holm", paired=TRUE)
# Tukey法。対応ありのデータでTukey法を使うのはだめ、と誰かが言ってた気がするが
require(multcomp)
summary(glht(aovres,linfct=mcp(x="Tukey"))) # aov関数の結果は受け取れず、エラーを返す
summary(glht(lmeres,linfct=mcp(x="Tukey")))
summary(glht(lmerres,linfct=mcp(x="Tukey")))
参考: http://gribblelab.org/2009/03/09/repeated-measures-anova-using-r/
愚痴: 分散分析というのはマイナーな手法なのか、解説がない。aovを使ったもの (タイプ1平方和) は多いが、タイプ3では計算できない。carパッケージのAnova関数を使えばタイプ3で実行できるが、今度は誤差項を指定できない。混合デザインに使えなくもないが。事後検定についても書いてあるところはないし、aov関数のように汎用性が高くないし、分析結果に変量効果 (random effect) が入らない (たぶん計算はしてる) 。
ほとんどのページがしれっとaovでのやり方を書いてるけどそれありなの?