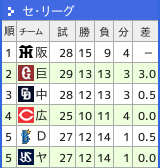
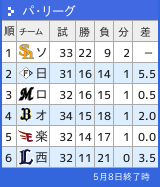
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
データフレームの1列を取り出してデータフレームにする
y <- iris[1]
y <- iris[2:4]
## y <- iris[,1]だとベクトルになる
## y <- iris[,2:4]はデータフレームになる
データフレームの一部を取り出してデータフレームにする
yy <- cbind(iris[1], iris[4:5], iris[1:2])
## カンマをつけても同じ yy <- cbind(iris[,1], iris[,4:5], iris[,1:2])
データフレームの1列を取り出してデータフレームにする
onedf <- data.frame("one"=c(df$xxxx))
# 名前をつけないと変数名が"df$xxx"になる
データフレームや行列を結合する
ndf <- cbind(onedf, onedf2)
# 変数名は元のデータフレームについている名前になる
ndf <- cbind(onedf$one, onedf2$one2)
# こうすると変数名がなくなってV1...とかが勝手につく
欠損値を除いたデータフレームをつくる
ndf <- na.omit(df)
ndf[,2] <- factor(ndf[,2])
## 0がカウントされている変数にfactor関数を使うと消える
y <- iris[1]
y <- iris[2:4]
## y <- iris[,1]だとベクトルになる
## y <- iris[,2:4]はデータフレームになる
データフレームの一部を取り出してデータフレームにする
yy <- cbind(iris[1], iris[4:5], iris[1:2])
## カンマをつけても同じ yy <- cbind(iris[,1], iris[,4:5], iris[,1:2])
データフレームの1列を取り出してデータフレームにする
onedf <- data.frame("one"=c(df$xxxx))
# 名前をつけないと変数名が"df$xxx"になる
データフレームや行列を結合する
ndf <- cbind(onedf, onedf2)
# 変数名は元のデータフレームについている名前になる
ndf <- cbind(onedf$one, onedf2$one2)
# こうすると変数名がなくなってV1...とかが勝手につく
欠損値を除いたデータフレームをつくる
ndf <- na.omit(df)
ndf[,2] <- factor(ndf[,2])
## 0がカウントされている変数にfactor関数を使うと消える
PR
データフレームを範囲指定して因子変数にする
Rjpwikiより
または
for (i in 4:24) {
datf[,i] <- factor(dat[,i]) # カンマの有無に注意
}
Rjpwikiより
datf[4:24] <- lapply(dat[4:24], factor)lapply(dat[4:24], factor)これはなんかうまくいかない
または
for (i in 4:24) {
datf[,i] <- factor(dat[,i]) # カンマの有無に注意
}
データフレーム全体で、変数名と型を調べ、縦に表示する
data.frame(1:ncol(iris), sapply(iris, class))
data.frame(1:ncol(iris), sapply(iris, class), colnames(iris))
##順序尺度の場合はうまく動作しない
データフレームの列や行の数を調べる
ncol(df) # 列数
nrow(df) # 行数
変数名を取り出す
colnames(df) # 変数名を取り出す。行はrownames(df)
因子変数の因子数 (カテゴリー数) を調べる
length(levels(iris$Species))
因子変数のカテゴリー名を調べる
levels(iris$Species)
[1] "setosa" "versicolor" "virginica"
順序尺度かどうか調べる
class(iris$Species)
[1] "factor"
# ただの因子はfactor, 順序があるとorderedになる。
# levelはただの並び順で順序はついていない (論理式は使えない)
順序尺度の順序を調べる
x <- ordered(iris$Species) # irisのSpeciesを順序づける
levels(x)
"setosa" "versicolor" "virginica"
小 => 大の順になっている。
summary(x)でもこの順で集計される
data.frame(1:ncol(iris), sapply(iris, class))
data.frame(1:ncol(iris), sapply(iris, class), colnames(iris))
##順序尺度の場合はうまく動作しない
データフレームの列や行の数を調べる
ncol(df) # 列数
nrow(df) # 行数
変数名を取り出す
colnames(df) # 変数名を取り出す。行はrownames(df)
因子変数の因子数 (カテゴリー数) を調べる
length(levels(iris$Species))
因子変数のカテゴリー名を調べる
levels(iris$Species)
[1] "setosa" "versicolor" "virginica"
順序尺度かどうか調べる
class(iris$Species)
[1] "factor"
# ただの因子はfactor, 順序があるとorderedになる。
# levelはただの並び順で順序はついていない (論理式は使えない)
順序尺度の順序を調べる
x <- ordered(iris$Species) # irisのSpeciesを順序づける
levels(x)
"setosa" "versicolor" "virginica"
小 => 大の順になっている。
summary(x)でもこの順で集計される
Rで変量要因を投入した分析を
行うにはlme4パッケージのlmer関数を使うらしい
これでランダム切片/傾きモデルを検討できる。
あと、glmmというのもあるらしい
…そのうち調べよう
行うにはlme4パッケージのlmer関数を使うらしい
これでランダム切片/傾きモデルを検討できる。
あと、glmmというのもあるらしい
…そのうち調べよう
aov関数の方がいろんな用途に使えて便利そうなのだが、タイプ1平方和を計算する。
なので、carパッケージのAnova関数を勉強しようと思っていた。
タイプIIかタイプIIIかは議論の分かれるところらしく、別に個人的にはタイプIIでもよいと思う。
しかし、上述のとおりaovではタイプIしか出ない…と思っていたのだが、基本に返って (今頃) Jonathan Baronの解説を読んでみると、SASやSPSSで平方和の指定をデフォルトのタイプ3からタイプ2にすれば、RとSASやSPSSは同じ結果になるよ、と書いてある。
「RでSASやSPSSと同じ出力が欲しい!」という人はそういう回答を求めてないと思うのだが…
ということは、非釣合型の分散分析でもaovでOKじゃないか、と思ったので上記HPの解説とanovakunのタイプ2指定の出力を比較してみた。
上記HP、6.8.1 Example 1: Mixed Models Approach to Within-Subject Factors (Hays, 1988, Table 13.21.2, p. 518)のUnbalanced (Nonorthogonal) Designs セクションより
以下転載
#データ生成
data1<-c(49,47,46,47,48,47,41,46,43,47,46,45,48,46,47,45,49,44,44,45,42,45,45,40,49,46,47,45,49,45,41,43,44,46,45,40,45,43,44,45,48,46,40,45,40,45,47,40)
### 交互作用ありにしたいときは以下を使う (適当にデータをいじったもの)
###data1<-c(49,10,46,47,48,47,41,46,43,5,46,5,48,46,47,45,100,44,44,45,42,45,45,40,49,46,47,45,49,45,41,43,44,46,100,40,45,43,44,45,48,46,40,45,10,45,10,40)
## 見やすくした (上記HPのコードを少し修正した。HPのままこぴぺするとエラーになる)
matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2")))
## anovakunでも検証するためにデータフレームにした
datkun <- data.frame(matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2"))))
# aov用のデータフレーム作成
Hays.df <- data.frame(rt = data1, subj = factor(rep(paste("subj", 1:12, sep=""), 4)), shape = factor(rep(rep(c("shape1", "shape2"), c(12, 12)), 2)), color = factor(rep(c("color1", "color2"), c(24, 24))))
# 非釣合型のデータにするため、grp変数を付け加える
Hays.df$grp <- factor(rep(c(1,1,1,1,1,1,1,1,2,2,2,2), 4))
## anovakun用のデータフレームにも付け加える
grp <- c(1,1,1,1,1,1,1,1,2,2,2,2)
datkun <- data.frame(cbind(grp, datkun))
# aovで分析
summary(aov(rt ~ grp*color*shape + Error(subj/(color+shape)), data=Hays.df))
## HPの記述は誤差項のカッコをつけわすれている; summary(aov(rt ~ grp*color*shape + Error(subj/shape+color), data=Hays.df))
# anovakunで分析
anovakun(datkun, "AsBC", 2, 2, 2, type2=T)
出力は全く同じになった。
結論: もうaovでやろう。lm, glmを使ったとき、気が向いたらAnovaを使おう。あと、そろそろまじめに平方和について勉強しよう
最近知ったのだが、anova関数はそもそも尤度を使ってモデル比較をするための関数らしい。知らないことが多いものだ
誤差項はスラッシュを使うよりも以下の形にしたほうが見やすい出力になる
被験者*対応がある要因をs:aとする。
後は足していくだけ
s:a + s:b + s:a:b
なので、carパッケージのAnova関数を勉強しようと思っていた。
タイプIIかタイプIIIかは議論の分かれるところらしく、別に個人的にはタイプIIでもよいと思う。
しかし、上述のとおりaovではタイプIしか出ない…と思っていたのだが、基本に返って (今頃) Jonathan Baronの解説を読んでみると、SASやSPSSで平方和の指定をデフォルトのタイプ3からタイプ2にすれば、RとSASやSPSSは同じ結果になるよ、と書いてある。
「RでSASやSPSSと同じ出力が欲しい!」という人はそういう回答を求めてないと思うのだが…
ということは、非釣合型の分散分析でもaovでOKじゃないか、と思ったので上記HPの解説とanovakunのタイプ2指定の出力を比較してみた。
上記HP、6.8.1 Example 1: Mixed Models Approach to Within-Subject Factors (Hays, 1988, Table 13.21.2, p. 518)のUnbalanced (Nonorthogonal) Designs セクションより
以下転載
#データ生成
data1<-c(49,47,46,47,48,47,41,46,43,47,46,45,48,46,47,45,49,44,44,45,42,45,45,40,49,46,47,45,49,45,41,43,44,46,45,40,45,43,44,45,48,46,40,45,40,45,47,40)
### 交互作用ありにしたいときは以下を使う (適当にデータをいじったもの)
###data1<-c(49,10,46,47,48,47,41,46,43,5,46,5,48,46,47,45,100,44,44,45,42,45,45,40,49,46,47,45,49,45,41,43,44,46,100,40,45,43,44,45,48,46,40,45,10,45,10,40)
## 見やすくした (上記HPのコードを少し修正した。HPのままこぴぺするとエラーになる)
matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2")))
## anovakunでも検証するためにデータフレームにした
datkun <- data.frame(matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2"))))
# aov用のデータフレーム作成
Hays.df <- data.frame(rt = data1, subj = factor(rep(paste("subj", 1:12, sep=""), 4)), shape = factor(rep(rep(c("shape1", "shape2"), c(12, 12)), 2)), color = factor(rep(c("color1", "color2"), c(24, 24))))
# 非釣合型のデータにするため、grp変数を付け加える
Hays.df$grp <- factor(rep(c(1,1,1,1,1,1,1,1,2,2,2,2), 4))
## anovakun用のデータフレームにも付け加える
grp <- c(1,1,1,1,1,1,1,1,2,2,2,2)
datkun <- data.frame(cbind(grp, datkun))
# aovで分析
summary(aov(rt ~ grp*color*shape + Error(subj/(color+shape)), data=Hays.df))
## HPの記述は誤差項のカッコをつけわすれている; summary(aov(rt ~ grp*color*shape + Error(subj/shape+color), data=Hays.df))
# anovakunで分析
anovakun(datkun, "AsBC", 2, 2, 2, type2=T)
出力は全く同じになった。
結論: もうaovでやろう。lm, glmを使ったとき、気が向いたらAnovaを使おう。あと、そろそろまじめに平方和について勉強しよう
最近知ったのだが、anova関数はそもそも尤度を使ってモデル比較をするための関数らしい。知らないことが多いものだ
誤差項はスラッシュを使うよりも以下の形にしたほうが見やすい出力になる
被験者*対応がある要因をs:aとする。
後は足していくだけ
s:a + s:b + s:a:b