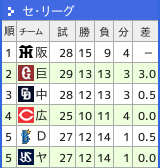
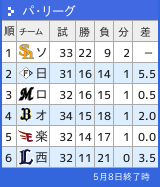
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
library(psych)
library(polycor)
library(foreign)
dat <- read.spss("http://www.indiana.edu/~statmath/stat/all/cfa/values_ord.sav", to.data.frame=T, max.value.labels=99)
pcres <- polychoric(dat)
print(pcres, digits=3)
# データをきちんと変換しておけばhetcor関数のほうが便利かも
x <- dat
dat2 <- data.frame(lapply(x, ordered))
sapply(dat2, class)
res1 <- polychoric(dat2) # エラー
res2 <- hetcor(dat2)
res2
res2$correlations # 相関行列だけとりだす
# 以下参考。感謝
# Rjpwiki: polycorパッケージによる順序相関係数の算出
# 小杉先生の順序尺度水準の相関係数について: http://www.kosugitti.net/labo/polynote.pdf
library(polycor)
library(foreign)
dat <- read.spss("http://www.indiana.edu/~statmath/stat/all/cfa/values_ord.sav", to.data.frame=T, max.value.labels=99)
pcres <- polychoric(dat)
print(pcres, digits=3)
# データをきちんと変換しておけばhetcor関数のほうが便利かも
x <- dat
dat2 <- data.frame(lapply(x, ordered))
sapply(dat2, class)
res1 <- polychoric(dat2) # エラー
res2 <- hetcor(dat2)
res2
res2$correlations # 相関行列だけとりだす
# 以下参考。感謝
# Rjpwiki: polycorパッケージによる順序相関係数の算出
# 小杉先生の順序尺度水準の相関係数について: http://www.kosugitti.net/labo/polynote.pdf
PR
psychパッケージのfa関数が不思議な挙動を見せるようなので
自分で調べてRjpwikiにレスしておいた。
せっかく調べたのでもう少し詳しく書いてみる
library(psych)
data(bfi)
x <- factor.pa(bfi, nfactors=3, rotate="promax")
# psych:::print.psych.faの中身を途中まで実行
# パラメータを設定しておく
digits = 2; all = FALSE; cut = NULL; sort = TRUE
#> psych:::print.psych.fa
#function (x, digits = 2, all = FALSE, cut = NULL, sort = FALSE,
# ...)
#{ ここまでは定義なので実行しないこと
if (!is.null(x$fn)) {
if (x$fn == "principal") {
cat("Principal Components Analysis")
}
else {
cat("Factor Analysis using method = ", x$fm)
}
}
cat("\nCall: ")
print(x$Call)
load <- x$loadings
if (is.null(cut))
cut <- 0
nitems <- dim(load)[1]
nfactors <- dim(load)[2]
loads <- data.frame(item = seq(1:nitems), cluster = rep(0,
nitems), unclass(load))
if (sort) {
loads$cluster <- apply(abs(load), 1, which.max)
ord <- sort(loads$cluster, index.return = TRUE)
loads[1:nitems, ] <- loads[ord$ix, ]
rownames(loads)[1:nitems] <- rownames(loads)[ord$ix]
items <- table(loads$cluster)
first <- 1
item <- loads$item
for (i in 1:length(items)) {
if (items[i] > 0) {
last <- first + items[i] - 1
ord <- sort(abs(loads[first:last, i + 2]), decreasing = TRUE,
index.return = TRUE)
loads[first:last, 3:(nfactors + 2)] <- load[item[ord$ix +
first - 1], ]
loads[first:last, 1] <- item[ord$ix + first -
1]
rownames(loads)[first:last] <- rownames(loads)[ord$ix +
first - 1]
first <- first + items[i]
}
}
}
if (cut > 0) {
ncol <- dim(loads)[2] - 2
rloads <- round(loads, digits)
fx <- format(rloads, digits = digits)
nc <- nchar(fx[1, 3], type = "c")
fx.1 <- fx[, 1, drop = FALSE]
fx.2 <- fx[, 3:(2 + ncol)]
load.2 <- as.matrix(loads[, 3:(ncol + 2)])
fx.2[abs(load.2) < cut] <- paste(rep(" ", nc), collapse = "")
fx <- data.frame(V = fx.1, fx.2)
if (dim(fx)[2] < 3)
colnames(fx) <- c("V", colnames(x$loadings))
if (nfactors > 1) {
if (is.null(x$Phi)) {
h2 <- rowSums(load.2^2)
}
else {
h2 <- diag(load.2 %*% x$Phi %*% t(load.2))
}
}
else {
h2 <- load.2^2
}
if (!is.null(x$uniquenesses)) {
u2 <- x$uniquenesses
}
else {
u2 <- (1 - h2)
}
vtotal <- sum(h2 + u2)
if (isTRUE(all.equal(vtotal, nitems))) {
cat("Standardized loadings based upon correlation matrix\n")
print(cbind(fx, h2, u2), quote = "FALSE", digits = digits)
}
else {
cat("Unstandardized loadings based upon covariance matrix\n")
print(cbind(fx, h2, u2, H2 = h2/(h2 + u2), U2 = u2/(h2 +
u2)), quote = "FALSE", digits = digits)
}
} else {
ncol <- dim(loads)[2] - 2
load.2 <- as.matrix(loads[, 3:(ncol + 2)])
if (nfactors > 1) {
if (is.null(x$Phi)) {
h2 <- rowSums(load.2^2)
}
else {
h2 <- diag(load.2 %*% x$Phi %*% t(load.2))
}
}
else {
h2 <- load.2^2
}
if (!is.null(x$uniquenesses)) {
u2 <- x$uniquenesses
}
else {
u2 <- (1 - h2)
}
vtotal <- sum(h2 + u2)
if (isTRUE(all.equal(vtotal, nitems))) {
cat("Standardized loadings based upon correlation matrix\n")
print(cbind(round(loads[-(1:2)], digits), h2, u2),
quote = "FALSE", digits = digits)
}
else {
cat("Unstandardized loadings based upon covariance matrix\n")
print(cbind(round(loads[-c(1:2)], digits), h2, u2,
H2 = h2/(h2 + u2), U2 = u2/(h2 + u2)), quote = "FALSE",
digits = digits)
}
}
if (is.null(x$Phi)) {
if (nfactors > 1) {
vx <- colSums(load.2^2)
}
else {
vx <- sum(load.2^2)
}
} else {
vx <- diag(x$Phi %*% t(load) %*% load)
}
names(vx) <- colnames(x$loadings)
varex <- rbind(`SS loadings` = vx)
varex <- rbind(varex, `Proportion Var` = vx/vtotal)
if (nfactors > 1)
varex <- rbind(varex, `Cumulative Var` = cumsum(vx/vtotal))
cat("\n")
print(round(varex, digits))
## この時点でh2はソートされているが、u2はソートされていない
h2
u2
# この後のコードでこの両者を使って標準化している (fx.2 ... sqrt(h2 + u2)) ため、誤りが生じます
#if (!isTRUE(all.equal(vtotal, nitems))) {
cat("\n Standardized loadings\n")
fx <- format(loads, digits = digits)
nc <- nchar(fx[1, 3], type = "c")
fx.1 <- fx[, 1, drop = FALSE]
fx.2 <- round(loads[, 3:(2 + ncol)]/sqrt(h2 + u2), digits)
########
# 誤った負荷量
fx.2
# 修正コード
u2 <- 1 - h2
fx.2 <- round(loads[, 3:(2 + ncol)]/sqrt(h2 + u2), digits)
fx.2
########
load.2 <- loads[, 3:(ncol + 2)]/sqrt(h2 + u2)
fx.2[abs(load.2) < cut] <- paste(rep(" ", nc), collapse = "")
fx <- data.frame(V = fx.1, fx.2)
## 以下本題
一エンドユーザーとしてみると、RがSPSSやSASより優れている点はフリーであること以外にない。Rを利用しているえらい人は (自分の本を売るためか) Rの使用をすすめる人が多いが、個人的には止めたほうがいいと思う。
それは使用に慣れるまでが大変で、容易に間違った結果を出力するからである。巷にあふれるRの入門書も誤った使用法を紹介しているものが山ほどある (厳密にいえば誤っているわけではなく、教科書の例題のようなごく限られた場合にのみ適用可能なコードを紹介している) …ような気がする。
特に、「世界中の人が使ってチェックするので,計算の信頼性もかなり高い。」といった宣伝文句はどうかと思う。baseに含まれている関数には当てはまるが、あまたあるパッケージの関数には当てはまらない。というか、下手に使うと間違った結果しか出力されず、信頼性はかなり低い。なぜなら、上記のようにフリーであるので誰にでも使用できるが、そのため誤ったコード例が量産されている (本サイトもかなり誤りがあるんです) 。エンドユーザーはコピペのように手軽に実行でき、しかも正しい結果の得られるものを求める。Rで自らのデータに合わせて適切にオプションを選択し、分析を実行していくのは容易ではない。
コピペで間違ったら自己責任でしょ、フリーなんだし、自分の研究なんだから…というのであれば、やはりSPSSやSASの方が優れているとしか思えない。
「Rで分析しました」という記述があったら、その結果を疑わしく見てしまうのは自分だけかな。「下手に使って、どっか間違ってんじゃないの?」と思ってしまう。
自分で調べてRjpwikiにレスしておいた。
せっかく調べたのでもう少し詳しく書いてみる
library(psych)
data(bfi)
x <- factor.pa(bfi, nfactors=3, rotate="promax")
# psych:::print.psych.faの中身を途中まで実行
# パラメータを設定しておく
digits = 2; all = FALSE; cut = NULL; sort = TRUE
#> psych:::print.psych.fa
#function (x, digits = 2, all = FALSE, cut = NULL, sort = FALSE,
# ...)
#{ ここまでは定義なので実行しないこと
if (!is.null(x$fn)) {
if (x$fn == "principal") {
cat("Principal Components Analysis")
}
else {
cat("Factor Analysis using method = ", x$fm)
}
}
cat("\nCall: ")
print(x$Call)
load <- x$loadings
if (is.null(cut))
cut <- 0
nitems <- dim(load)[1]
nfactors <- dim(load)[2]
loads <- data.frame(item = seq(1:nitems), cluster = rep(0,
nitems), unclass(load))
if (sort) {
loads$cluster <- apply(abs(load), 1, which.max)
ord <- sort(loads$cluster, index.return = TRUE)
loads[1:nitems, ] <- loads[ord$ix, ]
rownames(loads)[1:nitems] <- rownames(loads)[ord$ix]
items <- table(loads$cluster)
first <- 1
item <- loads$item
for (i in 1:length(items)) {
if (items[i] > 0) {
last <- first + items[i] - 1
ord <- sort(abs(loads[first:last, i + 2]), decreasing = TRUE,
index.return = TRUE)
loads[first:last, 3:(nfactors + 2)] <- load[item[ord$ix +
first - 1], ]
loads[first:last, 1] <- item[ord$ix + first -
1]
rownames(loads)[first:last] <- rownames(loads)[ord$ix +
first - 1]
first <- first + items[i]
}
}
}
if (cut > 0) {
ncol <- dim(loads)[2] - 2
rloads <- round(loads, digits)
fx <- format(rloads, digits = digits)
nc <- nchar(fx[1, 3], type = "c")
fx.1 <- fx[, 1, drop = FALSE]
fx.2 <- fx[, 3:(2 + ncol)]
load.2 <- as.matrix(loads[, 3:(ncol + 2)])
fx.2[abs(load.2) < cut] <- paste(rep(" ", nc), collapse = "")
fx <- data.frame(V = fx.1, fx.2)
if (dim(fx)[2] < 3)
colnames(fx) <- c("V", colnames(x$loadings))
if (nfactors > 1) {
if (is.null(x$Phi)) {
h2 <- rowSums(load.2^2)
}
else {
h2 <- diag(load.2 %*% x$Phi %*% t(load.2))
}
}
else {
h2 <- load.2^2
}
if (!is.null(x$uniquenesses)) {
u2 <- x$uniquenesses
}
else {
u2 <- (1 - h2)
}
vtotal <- sum(h2 + u2)
if (isTRUE(all.equal(vtotal, nitems))) {
cat("Standardized loadings based upon correlation matrix\n")
print(cbind(fx, h2, u2), quote = "FALSE", digits = digits)
}
else {
cat("Unstandardized loadings based upon covariance matrix\n")
print(cbind(fx, h2, u2, H2 = h2/(h2 + u2), U2 = u2/(h2 +
u2)), quote = "FALSE", digits = digits)
}
} else {
ncol <- dim(loads)[2] - 2
load.2 <- as.matrix(loads[, 3:(ncol + 2)])
if (nfactors > 1) {
if (is.null(x$Phi)) {
h2 <- rowSums(load.2^2)
}
else {
h2 <- diag(load.2 %*% x$Phi %*% t(load.2))
}
}
else {
h2 <- load.2^2
}
if (!is.null(x$uniquenesses)) {
u2 <- x$uniquenesses
}
else {
u2 <- (1 - h2)
}
vtotal <- sum(h2 + u2)
if (isTRUE(all.equal(vtotal, nitems))) {
cat("Standardized loadings based upon correlation matrix\n")
print(cbind(round(loads[-(1:2)], digits), h2, u2),
quote = "FALSE", digits = digits)
}
else {
cat("Unstandardized loadings based upon covariance matrix\n")
print(cbind(round(loads[-c(1:2)], digits), h2, u2,
H2 = h2/(h2 + u2), U2 = u2/(h2 + u2)), quote = "FALSE",
digits = digits)
}
}
if (is.null(x$Phi)) {
if (nfactors > 1) {
vx <- colSums(load.2^2)
}
else {
vx <- sum(load.2^2)
}
} else {
vx <- diag(x$Phi %*% t(load) %*% load)
}
names(vx) <- colnames(x$loadings)
varex <- rbind(`SS loadings` = vx)
varex <- rbind(varex, `Proportion Var` = vx/vtotal)
if (nfactors > 1)
varex <- rbind(varex, `Cumulative Var` = cumsum(vx/vtotal))
cat("\n")
print(round(varex, digits))
## この時点でh2はソートされているが、u2はソートされていない
h2
u2
# この後のコードでこの両者を使って標準化している (fx.2 ... sqrt(h2 + u2)) ため、誤りが生じます
#if (!isTRUE(all.equal(vtotal, nitems))) {
cat("\n Standardized loadings\n")
fx <- format(loads, digits = digits)
nc <- nchar(fx[1, 3], type = "c")
fx.1 <- fx[, 1, drop = FALSE]
fx.2 <- round(loads[, 3:(2 + ncol)]/sqrt(h2 + u2), digits)
########
# 誤った負荷量
fx.2
# 修正コード
u2 <- 1 - h2
fx.2 <- round(loads[, 3:(2 + ncol)]/sqrt(h2 + u2), digits)
fx.2
########
load.2 <- loads[, 3:(ncol + 2)]/sqrt(h2 + u2)
fx.2[abs(load.2) < cut] <- paste(rep(" ", nc), collapse = "")
fx <- data.frame(V = fx.1, fx.2)
## 以下本題
一エンドユーザーとしてみると、RがSPSSやSASより優れている点はフリーであること以外にない。Rを利用しているえらい人は (自分の本を売るためか) Rの使用をすすめる人が多いが、個人的には止めたほうがいいと思う。
それは使用に慣れるまでが大変で、容易に間違った結果を出力するからである。巷にあふれるRの入門書も誤った使用法を紹介しているものが山ほどある (厳密にいえば誤っているわけではなく、教科書の例題のようなごく限られた場合にのみ適用可能なコードを紹介している) …ような気がする。
特に、「世界中の人が使ってチェックするので,計算の信頼性もかなり高い。」といった宣伝文句はどうかと思う。baseに含まれている関数には当てはまるが、あまたあるパッケージの関数には当てはまらない。というか、下手に使うと間違った結果しか出力されず、信頼性はかなり低い。なぜなら、上記のようにフリーであるので誰にでも使用できるが、そのため誤ったコード例が量産されている (本サイトもかなり誤りがあるんです) 。エンドユーザーはコピペのように手軽に実行でき、しかも正しい結果の得られるものを求める。Rで自らのデータに合わせて適切にオプションを選択し、分析を実行していくのは容易ではない。
コピペで間違ったら自己責任でしょ、フリーなんだし、自分の研究なんだから…というのであれば、やはりSPSSやSASの方が優れているとしか思えない。
「Rで分析しました」という記述があったら、その結果を疑わしく見てしまうのは自分だけかな。「下手に使って、どっか間違ってんじゃないの?」と思ってしまう。
# 小塩先生のホームページからデータを拝借、出力を参照。感謝。
# http://psy.isc.chubu.ac.jp/~oshiolab/teaching_folder/datakaiseki_folder/add_folder/daad_01.html
ex <- c(3,6,6,6,5,4,6,5,6,6,5,5,6,5,5,6,4,6,5,4)
so <- c(4,6,5,7,7,5,6,5,6,5,4,5,6,5,6,6,4,6,3,6)
ac <- c(4,7,7,5,6,5,7,4,6,6,4,6,5,4,4,6,3,7,4,6)
il <- c(5,8,5,4,5,5,6,5,7,6,5,5,5,4,5,4,6,4,3,3)
re <- c(4,7,5,6,5,6,4,5,7,5,5,4,6,5,6,4,5,5,5,5)
fl <- c(4,7,6,5,5,6,4,6,6,5,5,5,5,3,6,5,6,5,4,4)
dat <- data.frame(ex, so, ac, il, re, fl)
library(psych)
fa.parallel(dat) # 固有値の推移。PC Actual Dataが主成分解 (だと思う) 。
fit <- principal(dat, nfactors=2, rotate=F, scores=T) # 固有値1を超えるのは2つなのでnfactorsで指定する。
print(fit, digits=3)
fit$scores
cor(fit$scores)
plot(fit) # 主成分得点からみる散布図
# 結論:psychパッケージは偉大である。
# http://psy.isc.chubu.ac.jp/~oshiolab/teaching_folder/datakaiseki_folder/add_folder/daad_01.html
ex <- c(3,6,6,6,5,4,6,5,6,6,5,5,6,5,5,6,4,6,5,4)
so <- c(4,6,5,7,7,5,6,5,6,5,4,5,6,5,6,6,4,6,3,6)
ac <- c(4,7,7,5,6,5,7,4,6,6,4,6,5,4,4,6,3,7,4,6)
il <- c(5,8,5,4,5,5,6,5,7,6,5,5,5,4,5,4,6,4,3,3)
re <- c(4,7,5,6,5,6,4,5,7,5,5,4,6,5,6,4,5,5,5,5)
fl <- c(4,7,6,5,5,6,4,6,6,5,5,5,5,3,6,5,6,5,4,4)
dat <- data.frame(ex, so, ac, il, re, fl)
library(psych)
fa.parallel(dat) # 固有値の推移。PC Actual Dataが主成分解 (だと思う) 。
fit <- principal(dat, nfactors=2, rotate=F, scores=T) # 固有値1を超えるのは2つなのでnfactorsで指定する。
print(fit, digits=3)
fit$scores
cor(fit$scores)
plot(fit) # 主成分得点からみる散布図
# 結論:psychパッケージは偉大である。
# 足立浩平 (2006). 多変量データ解析法――心理・教育・社会系のための入門―― ナカニシヤ出版の例より
# 因子分析と主成分分析は似て非なる方法であることに注意
# 以下のサイトを参考。というかRコマンダーでの例をそのままコードにしただけ。感謝
http://www.nanzan-u.ac.jp/~kamiya/r/content/rcmdrprin.html
http://www.statmethods.net/advstats/factor.html
# 表 3.1 p24
ppt <- c(88,52,77,35,60,97,48,66,78)
wrt <- c(70,78,87,40,43,95,62,66,50)
int <- c(65,88,89,43,40,91,83,65,48)
dat <- data.frame(ppt, wrt, int)
mean(dat)
sapply(dat, var)
fit <- princomp(~ppt+wrt+int, cor=F, data=dat) # 相関行列をもとにした分析をするにはcor=Tとする
fit2 <- prcomp(dat, cor=F)
# 主成分得点 表3.1 p22
# 単にprincomp(dat) でも同じ
fit$scores
fit2$x
# 各主成分の係数 表3.5 p32
loadings(fit)
unclass(loadings(fit)) # 行列にしただけ
fit2$rotation
# 固有ベクトルなので以下と同じ
eigen(cov(dat))$vectors
# 各主成分得点どうしの共分散 表3.6 p32
cov(fit$scores)
round(cov(fit$scores),1) # みやすく四捨五入
round(cov(fit2$x),1)
# 成分行列 (いわゆる負荷量)
t(fit$sd * t(fit$loadings ) )[, drop = FALSE]
t(fit2$sdev * t(fit2$rotation ) )[, drop = FALSE]
# 結果が異なる。おそらくprcompは Unlike princomp, variances are computed with the usual divisor N - 1. であるため、標準偏差の値が異なる
fit$sd
fit2$sdev
sqrt(fit2$sdev^2 *(8/9)) # 不偏分散を標本に戻すと一致
# 説明率、累積説明率 図3.6 p27
summary(fit)
print(summary(fit2), digits=10) #
基本的には同じ結果だが、prcompのsummaryは小数点第5桁までで区切っているのがわかる
# 固有値
fit$sd^2
fit2$sdev^2
# 固有値なので以下と同じ
eigen(cov(dat))$value
## おまけ。psychパッケージでもやってみる
fit2 <- princomp(~ppt+wrt+int, cor=T, data=dat) # 相関行列を使うよう指定してprincomp関数で実行
library(psych)
fit3 <- principal(dat, nfactors=3, rotate=F, scores=T)# psychパッケージでは標準化した相関行列がデフォルトで用いられる。回転もデフォルトだとバリマックス回転が行われる
summary(fit2) # princomp関数
print(fit3, digits=5) # psychパッケージのprincipal関数
fit2$scores
fit3$scores # psychパッケージでは標準化した主成分得点が算出される
## 覚書
prcomp関数は各個体の平均から偏差を求めるQモード法。対してprincomp, principal関数はRモード法。Qモード法は変数の数 > サンプルサイズ でも実行できる。
# 因子分析と主成分分析は似て非なる方法であることに注意
# 以下のサイトを参考。というかRコマンダーでの例をそのままコードにしただけ。感謝
http://www.nanzan-u.ac.jp/~kamiya/r/content/rcmdrprin.html
http://www.statmethods.net/advstats/factor.html
# 表 3.1 p24
ppt <- c(88,52,77,35,60,97,48,66,78)
wrt <- c(70,78,87,40,43,95,62,66,50)
int <- c(65,88,89,43,40,91,83,65,48)
dat <- data.frame(ppt, wrt, int)
mean(dat)
sapply(dat, var)
fit <- princomp(~ppt+wrt+int, cor=F, data=dat) # 相関行列をもとにした分析をするにはcor=Tとする
fit2 <- prcomp(dat, cor=F)
# 主成分得点 表3.1 p22
# 単にprincomp(dat) でも同じ
fit$scores
fit2$x
# 各主成分の係数 表3.5 p32
loadings(fit)
unclass(loadings(fit)) # 行列にしただけ
fit2$rotation
# 固有ベクトルなので以下と同じ
eigen(cov(dat))$vectors
# 各主成分得点どうしの共分散 表3.6 p32
cov(fit$scores)
round(cov(fit$scores),1) # みやすく四捨五入
round(cov(fit2$x),1)
# 成分行列 (いわゆる負荷量)
t(fit$sd * t(fit$loadings ) )[, drop = FALSE]
t(fit2$sdev * t(fit2$rotation ) )[, drop = FALSE]
# 結果が異なる。おそらくprcompは Unlike princomp, variances are computed with the usual divisor N - 1. であるため、標準偏差の値が異なる
fit$sd
fit2$sdev
sqrt(fit2$sdev^2 *(8/9)) # 不偏分散を標本に戻すと一致
# 説明率、累積説明率 図3.6 p27
summary(fit)
print(summary(fit2), digits=10) #
基本的には同じ結果だが、prcompのsummaryは小数点第5桁までで区切っているのがわかる
# 固有値
fit$sd^2
fit2$sdev^2
# 固有値なので以下と同じ
eigen(cov(dat))$value
## おまけ。psychパッケージでもやってみる
fit2 <- princomp(~ppt+wrt+int, cor=T, data=dat) # 相関行列を使うよう指定してprincomp関数で実行
library(psych)
fit3 <- principal(dat, nfactors=3, rotate=F, scores=T)# psychパッケージでは標準化した相関行列がデフォルトで用いられる。回転もデフォルトだとバリマックス回転が行われる
summary(fit2) # princomp関数
print(fit3, digits=5) # psychパッケージのprincipal関数
fit2$scores
fit3$scores # psychパッケージでは標準化した主成分得点が算出される
## 覚書
prcomp関数は各個体の平均から偏差を求めるQモード法。対してprincomp, principal関数はRモード法。Qモード法は変数の数 > サンプルサイズ でも実行できる。
# 心理統計学の基礎 共分散構造分析 p351-363 の例を実行
# ...してみようと思ったら以下のURLにそのものズバリが載っていた。感謝。自分用にコピペしておく
# http://home.hiroshima-u.ac.jp/ksatoh/documents/Rsem.txt
# http://home.hiroshima-u.ac.jp/ksatoh/download.htm
library(sem)
# 表10-8 観測変数間の相関係数 p354
mat <- matrix(c(
1.000,0.160,0.302,0.461,0.299,0.152,0.134,0.182,0.251,0.372,0.157,0.203,
0.160,1.000,0.341,0.400,0.404,0.320,0.403,0.374,0.285,0.100,0.291,-0.014,
0.302,0.341,1.000,0.372,0.552,0.476,0.467,0.572,0.316,0.408,0.393,0.369,
0.461,0.400,0.372,1.000,0.302,0.225,0.256,0.255,0.164,0.236,0.229,0.224,
0.299,0.404,0.552,0.302,1.000,0.708,0.623,0.776,0.361,0.294,0.472,0.342,
0.152,0.320,0.476,0.225,0.708,1.000,0.324,0.769,0.295,0.206,0.351,0.202,
0.134,0.403,0.467,0.256,0.623,0.324,1.000,0.724,0.260,0.071,0.204,0.152,
0.182,0.374,0.572,0.255,0.776,0.769,0.724,1.000,0.284,0.142,0.320,0.189,
0.251,0.285,0.316,0.164,0.361,0.295,0.260,0.284,1.000,0.295,0.290,0.418,
0.372,0.100,0.408,0.236,0.294,0.206,0.071,0.142,0.295,1.000,0.468,0.351,
0.157,0.291,0.393,0.229,0.472,0.351,0.204,0.320,0.290,0.468,1.000,0.385,
0.203,-0.014,0.369,0.224,0.342,0.202,0.152,0.189,0.418,0.351,0.385,1.000),
nr=12,
dimnames = list(paste("y", 1:12, sep=""), paste("y", 1:12, sep=""))
)
mat
# 日本語じゃなくて英語にしておく
model.agg <- specify.model()
mot -> y1, b11, NA
mot -> y2, b12, NA
mot -> y3, b13, NA
mot -> y4, b14, NA
int -> y5, NA, 1
int -> y6, b22, NA
int -> y7, b23, NA
int -> y8, b24, NA
agg -> y9, NA, 1
agg -> y10, b32, NA
agg -> y11, b33, NA
agg -> y12, b34, NA
mot -> int, g11, NA
mot -> agg, g12, NA
y1 <-> y1, e1, NA
y2 <-> y2, e2, NA
y3 <-> y3, e3, NA
y4 <-> y4, e4, NA
y5 <-> y5, e5, NA
y6 <-> y6, e6, NA
y7 <-> y7, e7, NA
y8 <-> y8, e8, NA
y9 <-> y9, e9, NA
y10 <-> y10, e10, NA
y11 <-> y11, e11, NA
y12 <-> y12, e12, NA
mot <-> mot, NA, 1
int <-> int, delta2, NA
agg <-> agg, delta3, NA
sem.agg <- sem(model.agg, mat, N=50)
summary(sem.agg)
std.coef(sem.agg,digit=4) # 標準解 図10-6と一致するのはこっち
# 図10-6 くらいは自分で書いてみよう
library(Rgraphviz)
path.diagram(sem.agg, ignore.double=FALSE, edge.labels="values", digits=3, file="out2")
# やはり、日本語フォントでは表示できない。Graphvizのバージョンが古いのかもしれない
# しかたないので協調性はagg, 母親価値はmot, 相互作用経験はintとした。でも文字の一部が消えたり、どうもうまくいかない。
# そのうえ、sem.aggの係数で描画するため、標準解を出力できない
# ...してみようと思ったら以下のURLにそのものズバリが載っていた。感謝。自分用にコピペしておく
# http://home.hiroshima-u.ac.jp/ksatoh/documents/Rsem.txt
# http://home.hiroshima-u.ac.jp/ksatoh/download.htm
library(sem)
# 表10-8 観測変数間の相関係数 p354
mat <- matrix(c(
1.000,0.160,0.302,0.461,0.299,0.152,0.134,0.182,0.251,0.372,0.157,0.203,
0.160,1.000,0.341,0.400,0.404,0.320,0.403,0.374,0.285,0.100,0.291,-0.014,
0.302,0.341,1.000,0.372,0.552,0.476,0.467,0.572,0.316,0.408,0.393,0.369,
0.461,0.400,0.372,1.000,0.302,0.225,0.256,0.255,0.164,0.236,0.229,0.224,
0.299,0.404,0.552,0.302,1.000,0.708,0.623,0.776,0.361,0.294,0.472,0.342,
0.152,0.320,0.476,0.225,0.708,1.000,0.324,0.769,0.295,0.206,0.351,0.202,
0.134,0.403,0.467,0.256,0.623,0.324,1.000,0.724,0.260,0.071,0.204,0.152,
0.182,0.374,0.572,0.255,0.776,0.769,0.724,1.000,0.284,0.142,0.320,0.189,
0.251,0.285,0.316,0.164,0.361,0.295,0.260,0.284,1.000,0.295,0.290,0.418,
0.372,0.100,0.408,0.236,0.294,0.206,0.071,0.142,0.295,1.000,0.468,0.351,
0.157,0.291,0.393,0.229,0.472,0.351,0.204,0.320,0.290,0.468,1.000,0.385,
0.203,-0.014,0.369,0.224,0.342,0.202,0.152,0.189,0.418,0.351,0.385,1.000),
nr=12,
dimnames = list(paste("y", 1:12, sep=""), paste("y", 1:12, sep=""))
)
mat
# 日本語じゃなくて英語にしておく
model.agg <- specify.model()
mot -> y1, b11, NA
mot -> y2, b12, NA
mot -> y3, b13, NA
mot -> y4, b14, NA
int -> y5, NA, 1
int -> y6, b22, NA
int -> y7, b23, NA
int -> y8, b24, NA
agg -> y9, NA, 1
agg -> y10, b32, NA
agg -> y11, b33, NA
agg -> y12, b34, NA
mot -> int, g11, NA
mot -> agg, g12, NA
y1 <-> y1, e1, NA
y2 <-> y2, e2, NA
y3 <-> y3, e3, NA
y4 <-> y4, e4, NA
y5 <-> y5, e5, NA
y6 <-> y6, e6, NA
y7 <-> y7, e7, NA
y8 <-> y8, e8, NA
y9 <-> y9, e9, NA
y10 <-> y10, e10, NA
y11 <-> y11, e11, NA
y12 <-> y12, e12, NA
mot <-> mot, NA, 1
int <-> int, delta2, NA
agg <-> agg, delta3, NA
sem.agg <- sem(model.agg, mat, N=50)
summary(sem.agg)
std.coef(sem.agg,digit=4) # 標準解 図10-6と一致するのはこっち
# 図10-6 くらいは自分で書いてみよう
library(Rgraphviz)
path.diagram(sem.agg, ignore.double=FALSE, edge.labels="values", digits=3, file="out2")
# やはり、日本語フォントでは表示できない。Graphvizのバージョンが古いのかもしれない
# しかたないので協調性はagg, 母親価値はmot, 相互作用経験はintとした。でも文字の一部が消えたり、どうもうまくいかない。
# そのうえ、sem.aggの係数で描画するため、標準解を出力できない