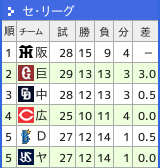
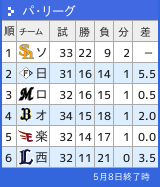
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
# 心理統計学の基礎, p347-350のもの。感謝
# 表10-1 p318 相関行列
mat <- matrix(c(
1.000,0.033,0.315,0.456,0.266,0.607,0.228,0.419,
0.033,1.000,0.637,0.250,0.528,0.195,0.522,0.420,
0.315,0.637,1.000,0.333,0.880,0.237,0.750,0.328,
0.456,0.250,0.333,1.000,0.362,0.432,0.398,0.449,
0.266,0.528,0.880,0.362,1.000,0.252,0.738,0.269,
0.607,0.195,0.237,0.432,0.252,1.000,0.335,0.463,
0.228,0.522,0.750,0.398,0.738,0.335,1.000,0.238,
0.419,0.420,0.328,0.449,0.269,0.463,0.238,1.000),
nrow=8, dimnames=list(c("onw", "yok", "gai", "sin", "sha", "kyo", "sek", "sun"), c("onw", "yok", "gai", "sin", "sha", "kyo", "sek", "sun"))
)
library(sem)
model <- specify.model()
f1 -> yok, b12, 1
f1 -> gai, b13, NA
f1 -> sha, b15, NA
f1 -> sek, b17, NA
f2 -> onw, b21, NA
f2 -> sin, b24, NA
f2 -> kyo, b26, NA
f2 -> sun, b28, NA
f1 <-> f2, g11, NA
f1 <-> f1, NA, 1
f2 <-> f2, NA,1
onw <-> onw, e1, NA
yok <-> yok, e2, NA
gai <-> gai, e3, NA
sin <-> sin, e4, NA
sha <-> sha, e5, NA
kyo <-> kyo, e6, NA
sek <-> sek, e7, NA
sun <-> sun, e8, NA
semres <- sem(model, mat, N=200)
# 表10-7, p394 のものと一致
summary(semres)
# 適合度指標基準の覚書
# 以下参考にしたサイト。感謝。David Kennyのをちゃんと読むのが一番いいだろう
# David Kenny
# 小塩先生のページ。感謝
# Rでsem入門。
# Indiana Universityとpdf
# 荘島先生のサイト
# GFI, AGFIは.90以上。サンプルサイズが大きいときは無視
# RMSEAは小さいほうがいい。0.05未満
# AIC, BICは相対基準。小さいほうがいい
# 表10-1 p318 相関行列
mat <- matrix(c(
1.000,0.033,0.315,0.456,0.266,0.607,0.228,0.419,
0.033,1.000,0.637,0.250,0.528,0.195,0.522,0.420,
0.315,0.637,1.000,0.333,0.880,0.237,0.750,0.328,
0.456,0.250,0.333,1.000,0.362,0.432,0.398,0.449,
0.266,0.528,0.880,0.362,1.000,0.252,0.738,0.269,
0.607,0.195,0.237,0.432,0.252,1.000,0.335,0.463,
0.228,0.522,0.750,0.398,0.738,0.335,1.000,0.238,
0.419,0.420,0.328,0.449,0.269,0.463,0.238,1.000),
nrow=8, dimnames=list(c("onw", "yok", "gai", "sin", "sha", "kyo", "sek", "sun"), c("onw", "yok", "gai", "sin", "sha", "kyo", "sek", "sun"))
)
library(sem)
model <- specify.model()
f1 -> yok, b12, 1
f1 -> gai, b13, NA
f1 -> sha, b15, NA
f1 -> sek, b17, NA
f2 -> onw, b21, NA
f2 -> sin, b24, NA
f2 -> kyo, b26, NA
f2 -> sun, b28, NA
f1 <-> f2, g11, NA
f1 <-> f1, NA, 1
f2 <-> f2, NA,1
onw <-> onw, e1, NA
yok <-> yok, e2, NA
gai <-> gai, e3, NA
sin <-> sin, e4, NA
sha <-> sha, e5, NA
kyo <-> kyo, e6, NA
sek <-> sek, e7, NA
sun <-> sun, e8, NA
semres <- sem(model, mat, N=200)
# 表10-7, p394 のものと一致
summary(semres)
# 適合度指標基準の覚書
# 以下参考にしたサイト。感謝。David Kennyのをちゃんと読むのが一番いいだろう
# David Kenny
# 小塩先生のページ。感謝
# Rでsem入門。
# Indiana Universityとpdf
# 荘島先生のサイト
# GFI, AGFIは.90以上。サンプルサイズが大きいときは無視
# RMSEAは小さいほうがいい。0.05未満
# AIC, BICは相対基準。小さいほうがいい
PR
# 狩野裕・三浦麻子 (2002). AMOS, EQS, CALISによるグラフィカル多変量解析――目で見る共分散構造分析 第2版 現代数学者
# p2, 表1のデータより
kk <- c(89,99,128,98,52,47,40,39,38,48,27,23) # 価格
sk <- c(4.3,1.9,5.2,5.1,4.0,4.8,8.7,8.2,3.3,3.9,8.2,7.2) # 走行距離
jn <- c(5,4,2,3,6,8,7,7,10,6,8,8) # 乗車年数
skn <- c(24,18,13,4,15,24,3,6,14,0,24,24) # 車検
dat <- data.frame(kk, sk, jn, skn)
dat2 <- scale(dat)
summary(lm(kk~jn))
library(lavaan) # 残念ながら、相関行列をデータとしてうけとることはできないらしい
# p15、パス解析モデル1
model1 <- '
sk ~ jn
kk ~ sk + skn
'
model1sc <- '
sk ~ jn
kk ~ sk + skn
'
# p16、パス解析モデル2
model2 <- '
sk ~ jn
kk ~ sk + skn + jn
'
model2sc <- '
sk ~ jn
kk ~ sk + skn + jn
'
fit1 <- sem(model1, data=dat)
fit1sc <- sem(model1sc, data=dat2)
fit2 <- sem(model2, data=dat)
fit2sc <- sem(model2sc, data=dat2)
summary(fit1)
summary(fit1sc)
summary(fit2)
summary(fit2sc)
参考
Rosseel, Y. lavaan: an R package for structural equation modeling and more
# p2, 表1のデータより
kk <- c(89,99,128,98,52,47,40,39,38,48,27,23) # 価格
sk <- c(4.3,1.9,5.2,5.1,4.0,4.8,8.7,8.2,3.3,3.9,8.2,7.2) # 走行距離
jn <- c(5,4,2,3,6,8,7,7,10,6,8,8) # 乗車年数
skn <- c(24,18,13,4,15,24,3,6,14,0,24,24) # 車検
dat <- data.frame(kk, sk, jn, skn)
dat2 <- scale(dat)
summary(lm(kk~jn))
library(lavaan) # 残念ながら、相関行列をデータとしてうけとることはできないらしい
# p15、パス解析モデル1
model1 <- '
sk ~ jn
kk ~ sk + skn
'
model1sc <- '
sk ~ jn
kk ~ sk + skn
'
# p16、パス解析モデル2
model2 <- '
sk ~ jn
kk ~ sk + skn + jn
'
model2sc <- '
sk ~ jn
kk ~ sk + skn + jn
'
fit1 <- sem(model1, data=dat)
fit1sc <- sem(model1sc, data=dat2)
fit2 <- sem(model2, data=dat)
fit2sc <- sem(model2sc, data=dat2)
summary(fit1)
summary(fit1sc)
summary(fit2)
summary(fit2sc)
参考
Rosseel, Y. lavaan: an R package for structural equation modeling and more
libcdt-4.dllがない などと言われて放置していたRgraphvizのインストール
こちらのページとこちらのページを参考にした。感謝
以下自分の覚書 (ほぼ上のリンクのコピペ)
1. http://www.graphviz.org/pub/graphviz/stable/windows/ より、graphviz-2.20.3a.msiをダウンロードしてインストール
## 以下、WindowsVistaでの設定
2. コントロールパネル -> システム -> システムの詳細設定 -> 詳細設定タブ -> 環境変数ボタン クリック
3. "システム環境変数" (下側) で"新規" をクリック
4. "変数名" には Path
"変数値" には C:\Program Files\Graphviz2.20\bin\
と入力。その後OK連打
5. コンピュータを再起動
## WindowsVistaの設定終了
6. Rを起動。コンソールに以下をコピペしてRgraphvizをインストール
source("http://bioconductor.org/biocLite.R");biocLite("Rgraphviz")
## ためしてみよう
library("Rgraphviz")
set.seed(123)
V <- letters[1:10]
M <- 1:4
g1 <- randomGraph(V, M, 0.2)
g1 <- layoutGraph(g1)
renderGraph(g1)
## 以下、インストールできた環境
> sessionInfo()
R version 2.11.1 (2010-05-31)
i386-pc-mingw32
locale:
[1] LC_COLLATE=Japanese_Japan.932 LC_CTYPE=Japanese_Japan.932
[3] LC_MONETARY=Japanese_Japan.932 LC_NUMERIC=C
[5] LC_TIME=Japanese_Japan.932
attached base packages:
[1] grid stats graphics grDevices utils datasets methods base
other attached packages:
[1] Rgraphviz_1.26.0 graph_1.26.0
loaded via a namespace (and not attached):
[1] tools_2.11.1
Rで確認的因子分析を行う。
Indiana Universityのサイトとデータを参考にする。感謝。
library(foreign)
dat <- data.frame(read.spss("http://www.indiana.edu/~statmath/stat/all/cfa/values.sav"))
library(sem)
model <- specify.model()
economic -> privtown, p1, 1
economic -> govtresp, p2, NA
economic -> compete, p3, NA
moral -> homosex, p4, NA
moral -> abortion, p5, NA
moral -> euthanas, p6, NA
privtown <-> privtown, e1, NA
govtresp <-> govtresp, e2, NA
compete <-> compete, e3, NA
homosex <-> homosex, e4, NA
abortion <-> abortion, e5, NA
euthanas <-> euthanas, e6, NA
economic <-> economic, NA, 1
moral <-> moral, NA, 1
economic <-> moral, em, NA
semres <- sem(model, cov(dat), nrow(dat))
(sumsem <- summary(semres))
std.coef(semres) # 標準化係数
# このサイトとこのサイトにある関数を利用してAIC, BIC, R二乗を出す。感謝
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1266106324")
sem.aic(semres) # AIC
rsquare.sem(semres) # R二乗
sumsem$chisq - sumsem$df * log(semres$N) # BIC
## 作図。Rgraphvizが上手くインストールできないので (libcdt-4.dllがないとかいわれる) のでGraphvizで書く
# とりあえずインストールはできた
# 以下のコードで出力されたdigraph...のテキストをGraphvizで描画する。Graphvizを起動し、File -> Newで出たウィンドウにコピペしてF5を押す。設定ダイアログは何もせずにOKとした
path.diagram(semres, ignore.double=FALSE, edge.labels="values", digits=3, standardize=T) # file="filename"と指定するとpdfが作業ディレクトリに出力される。だけど表示がおかしい
出力されたテキスト
# 望んだ作図ではないが、今日はこの辺で勘弁してやる
元サイトと全然結果が一致しない…まいったのう
## specify.model の覚書
変数間の関係、共分散・係数の名前、初期値
変数間の関係は -> か <-> で指定。前後に変数名を入れる
sem関数に与える相関・共分散行列にない名前は潜在変数になる
その次は共分散の名前。p1とか文字列で指定する。
その次が初期値。NAこれを推定する。
一方向 -> は回帰係数
両方向 <-> で変数名が違う場合は共分散
両方向で変数名が同じ場合はその変数の分散
係数をNAとすると固定母数として扱われる
潜在変数 (因子) の分散は固定母数1にすること
F1 <-> F1, NA, 1
観測変数の分散はNAにする
O1 <-> O1, e1, NA
同じ名前のパスが2つあると同値制約
同じ潜在変数同士 F1 <-F1 は負荷量にNA, 初期値に1を与えて固定母数にする
初心者の方へ: モデル式中にはパス係数、分散、共分散の全てを表記しなければならない。パス係数だけ指定してもモデルが推定されないので、要注意である (Rjiwikiより)
psychパッケージのstructure.sem関数でもモデルが書ける
Indiana Universityのサイトとデータを参考にする。感謝。
library(foreign)
dat <- data.frame(read.spss("http://www.indiana.edu/~statmath/stat/all/cfa/values.sav"))
library(sem)
model <- specify.model()
economic -> privtown, p1, 1
economic -> govtresp, p2, NA
economic -> compete, p3, NA
moral -> homosex, p4, NA
moral -> abortion, p5, NA
moral -> euthanas, p6, NA
privtown <-> privtown, e1, NA
govtresp <-> govtresp, e2, NA
compete <-> compete, e3, NA
homosex <-> homosex, e4, NA
abortion <-> abortion, e5, NA
euthanas <-> euthanas, e6, NA
economic <-> economic, NA, 1
moral <-> moral, NA, 1
economic <-> moral, em, NA
semres <- sem(model, cov(dat), nrow(dat))
(sumsem <- summary(semres))
std.coef(semres) # 標準化係数
# このサイトとこのサイトにある関数を利用してAIC, BIC, R二乗を出す。感謝
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1266106324")
sem.aic(semres) # AIC
rsquare.sem(semres) # R二乗
sumsem$chisq - sumsem$df * log(semres$N) # BIC
## 作図。
# とりあえずインストールはできた
# 以下のコードで出力されたdigraph...のテキストをGraphvizで描画する。Graphvizを起動し、File -> Newで出たウィンドウにコピペしてF5を押す。設定ダイアログは何もせずにOKとした
path.diagram(semres, ignore.double=FALSE, edge.labels="values", digits=3, standardize=T) # file="filename"と指定するとpdfが作業ディレクトリに出力される。だけど表示がおかしい
出力されたテキスト
# 望んだ作図ではないが、今日はこの辺で勘弁してやる
元サイトと全然結果が一致しない…まいったのう
## specify.model の覚書
変数間の関係、共分散・係数の名前、初期値
変数間の関係は -> か <-> で指定。前後に変数名を入れる
sem関数に与える相関・共分散行列にない名前は潜在変数になる
その次は共分散の名前。p1とか文字列で指定する。
その次が初期値。NAこれを推定する。
一方向 -> は回帰係数
両方向 <-> で変数名が違う場合は共分散
両方向で変数名が同じ場合はその変数の分散
係数をNAとすると固定母数として扱われる
潜在変数 (因子) の分散は固定母数1にすること
F1 <-> F1, NA, 1
観測変数の分散はNAにする
O1 <-> O1, e1, NA
同じ名前のパスが2つあると同値制約
同じ潜在変数同士 F1 <-F1 は負荷量にNA, 初期値に1を与えて固定母数にする
初心者の方へ: モデル式中にはパス係数、分散、共分散の全てを表記しなければならない。パス係数だけ指定してもモデルが推定されないので、要注意である (Rjiwikiより)
psychパッケージのstructure.sem関数でもモデルが書ける
固有値とスクリープロット
library(psych)
faev <- fa.parallel(dat3)
faev
因子推定
# 最尤法
factMLres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="ml", scores=T)
# 主因子法
factPAres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="pa", scores=T)
# 一般化最小二乗法
factGLSres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="gls", scores=T)
# 重みつき最小二乗法
factWLSres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="wls", scores=T)
# 最小残差法 (重みづけない最小二乗法)
factOLSres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="minres", scores=T)
# 結果の出力
print(factMLres, cut=0, sort=T, digits=5)
回転 (rotate="") のオプション
# 回転なし。いわゆる初期解ってやつ
"none"
# 直交解
"varimax", "quartimax", "bentlerT", and "geominT"
# 斜交解
"promax", "oblimin", "simplimax", "bentlerQ, and "geominQ" or "cluster"
因子得点はscores=T。推定は (たぶん) 回帰法
library(psych)
? fa
のDetailも勉強になる
library(psych)
faev <- fa.parallel(dat3)
faev
因子推定
# 最尤法
factMLres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="ml", scores=T)
# 主因子法
factPAres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="pa", scores=T)
# 一般化最小二乗法
factGLSres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="gls", scores=T)
# 重みつき最小二乗法
factWLSres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="wls", scores=T)
# 最小残差法 (重みづけない最小二乗法)
factOLSres <- fa(r=dat3, nfactors=3 ,rotate="promax", fm="minres", scores=T)
# 結果の出力
print(factMLres, cut=0, sort=T, digits=5)
回転 (rotate="") のオプション
# 回転なし。いわゆる初期解ってやつ
"none"
# 直交解
"varimax", "quartimax", "bentlerT", and "geominT"
# 斜交解
"promax", "oblimin", "simplimax", "bentlerQ, and "geominQ" or "cluster"
因子得点はscores=T。推定は (たぶん) 回帰法
library(psych)
? fa
のDetailも勉強になる