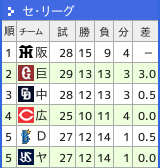
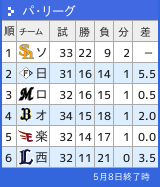
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
library(FactoMineR)
data (poison)
dat <- poison[,-c(1,2)]
# 各変数の度数
sapply(dat, table)
MCres <- MCA(dat, graph=F)
MCres
MCres$eig # 固有値と説明率
MCres$var$coord[,1:2] # カテゴリースコア。spssだと重心座標。符号が入れ替わる
MCres$var$eta2 # 判別測定
colSums(MCres$var$eta2) # 判別測定の合計
MCres$ind$coord[,1:2] # 個人スコア。spssとは結構違う。
# カテゴリースコアで数量化された
# 個人プロット
plot.MCA(MCres,invisible=c("var","quali.sup"))
MCres$ind$coord[23,1:2]
# カテゴリープロット
plot.MCA(MCres,invisible="ind")
# バイプロット
plot.MCA(MCres)
# 個体スコアについて
x <- as.vector(as.matrix(dat[1,]))
cs <- MCres$var$coord[,1:2] # カテゴリースコア
dm <- colSums(MCres$var$eta2) # 判別測定の合計
# 個体1のカテゴリースコア
cs[x[1],]
cs[x[2],]
cs[x[3],]
# 全部足す
(cs[x[1],]+cs[x[2],]+cs[x[3],])/dm[1:2] # spssではこれ
MCres$ind$coord[1,1:2]
# 個体スコアの計算方法が異なることがわかる
# そろそろspssを気にしないことにしよう
PR
Comment
Trackback
Trackback URL
Comment form