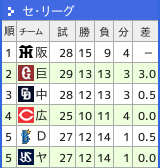
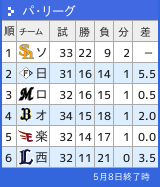
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
PR
データフレームdfの1列目がidv, 2列目がmod (以上独立変数)
4列目がdv (従属変数) として
以下は全て同じ結果を返す。列番号指定のときはas.matrixをつけないとダメ
ただし、個々の独立変数のF値等を見たいときは一番上
lm(dv ~ idv + mod, data = df)
lm(df[, 4] ~ as.matrix(df[c(1, 2)]))
lm(df[, 4] ~ as.matrix(df[, 1:2]))
lm(df[, 4] ~ as.matrix(df[1:2]))
結論:面倒でも独立変数は全部変数名と"+"でつなごう
4列目がdv (従属変数) として
以下は全て同じ結果を返す。列番号指定のときはas.matrixをつけないとダメ
ただし、個々の独立変数のF値等を見たいときは一番上
lm(dv ~ idv + mod, data = df)
lm(df[, 4] ~ as.matrix(df[c(1, 2)]))
lm(df[, 4] ~ as.matrix(df[, 1:2]))
lm(df[, 4] ~ as.matrix(df[1:2]))
結論:面倒でも独立変数は全部変数名と"+"でつなごう
大本。idv, mod, dv、全て連続変量
result <- lm(dv ~ idv*mod, df)
# これでidv, modの主効果、idv*modの交互作用項を検定している
# 結果
## 残差、偏回帰係数、その標準誤差、t値、p値、決定係数、調整済み決定係数、回帰式のF値
summary(result)
## QuantPsycパッケージのlm.beta関数で標準化偏回帰係数を出してくれるようだ
lm.beta(result)
## 各独立変数の平方和とF値
anova(result)
## VIFとトレランス
x <- df$idv*df$mod # 交互作用項をつくっておく
ndf <- cbind("idv"=c(df$idv), "mod"=c(df$mod), x) # 独立変数だけのデータフレームをつくっておく
diag(solve(cor(ndf))) # VIF計算
1/diag(solve(cor(ndf))) # トレランス
## モデルの比較
result2 <- lm(dv ~ idv+mod, df) # 交互作用なしモデル
anova(result2, result)
simple slope (単純傾斜)
# QuantPsycパッケージを利用する
## この関数の結果はSPSSによるものと一致しない。さらにanovaの分散分析表も一致しない。たぶん平方和の問題じゃないかと思う。別に一致する必要はないが...
library(QuantPsyc)
mresult <- moderate.lm(idv, mod, dv, df, mc = TRUE)
## mcはデータを中心化しているかどうか
## mresultは上記のresultと一致する
sresult <- sim.slopes(mresult, df$mod, zsd=1, mcz=FALSE)
## zsdはsimple slopeをつくるとき、SDをいくつとるか。普通は1SD (だったと思う)
sresult # 結果の出力。各群の切片、偏回帰係数、標準誤差、信頼区間が出力される
graph.mod(sresult, idv, dv, df, title = "Sslope", xlab = "Centered X", ylab = "Raw Y", ylimit = 1.5)
result <- lm(dv ~ idv*mod, df)
# これでidv, modの主効果、idv*modの交互作用項を検定している
# 結果
## 残差、偏回帰係数、その標準誤差、t値、p値、決定係数、調整済み決定係数、回帰式のF値
summary(result)
## QuantPsycパッケージのlm.beta関数で標準化偏回帰係数を出してくれるようだ
lm.beta(result)
## 各独立変数の平方和とF値
anova(result)
## VIFとトレランス
x <- df$idv*df$mod # 交互作用項をつくっておく
ndf <- cbind("idv"=c(df$idv), "mod"=c(df$mod), x) # 独立変数だけのデータフレームをつくっておく
diag(solve(cor(ndf))) # VIF計算
1/diag(solve(cor(ndf))) # トレランス
## モデルの比較
result2 <- lm(dv ~ idv+mod, df) # 交互作用なしモデル
anova(result2, result)
simple slope (単純傾斜)
# QuantPsycパッケージを利用する
## この関数の結果はSPSSによるものと一致しない。さらにanovaの分散分析表も一致しない。たぶん平方和の問題じゃないかと思う。別に一致する必要はないが...
library(QuantPsyc)
mresult <- moderate.lm(idv, mod, dv, df, mc = TRUE)
## mcはデータを中心化しているかどうか
## mresultは上記のresultと一致する
sresult <- sim.slopes(mresult, df$mod, zsd=1, mcz=FALSE)
## zsdはsimple slopeをつくるとき、SDをいくつとるか。普通は1SD (だったと思う)
sresult # 結果の出力。各群の切片、偏回帰係数、標準誤差、信頼区間が出力される
graph.mod(sresult, idv, dv, df, title = "Sslope", xlab = "Centered X", ylab = "Raw Y", ylimit = 1.5)