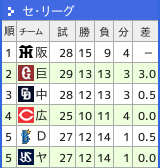
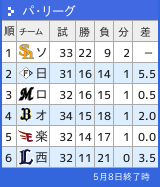
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
正準判別分析をやってみた3
#lda, candisでは判別が有意かどうかわからないのでちょっと調べてみたがmanova関数で検定していた。これで出力のWilksのところを見ればいいらしいが…ホンマ?
X<-as.matrix(iris[-5]) #データはRに入っているiris
manova.res<-manova(X~iris$Species)
print(summary(manova.res, test="Wilks") )
# 他の検定も可能
print(summary(manova.res, test="Pillai") )
print(summary(manova.res, test="Hotelling-Lawley") )
print(summary(manova.res, test="Roy") )
#lda, candisでは判別が有意かどうかわからないのでちょっと調べてみたがmanova関数で検定していた。これで出力のWilksのところを見ればいいらしいが…ホンマ?
X<-as.matrix(iris[-5]) #データはRに入っているiris
manova.res<-manova(X~iris$Species)
print(summary(manova.res, test="Wilks") )
# 他の検定も可能
print(summary(manova.res, test="Pillai") )
print(summary(manova.res, test="Hotelling-Lawley") )
print(summary(manova.res, test="Roy") )
PR
正準判別分析をやってみた2
(正準判別分析をやってみた1)
データはエヴェリット (2007) の頭蓋骨データ
青木先生のcandis関数
rescandis <- candis(skulls[2:5],skulls[1])
# 結果print(rescandis)
基本はhttp://aoki2.si.gunma-u.ac.jp/R/candis.htmlを参照
lda関数、エヴェリット (2007) と関連するところだけ列挙
判別係数 #ldaのCoefficients of linear discriminants、エヴェリット (2007) の正準係数
axis 1 axis 2 axis 3 axis 4
MB -0.126676 0.038738 0.092768 -0.14883986
- 中略 -
NH -0.082851 -0.077293 -0.294589 -0.06685888
constant 2.283887 -22.530493 4.409749 36.07126158
判別結果 #エヴェリット (2007) のプラグイン推定による分類表
classification
group
c1850BC c200BC c3300BC c4000BC cAD150
c1850BC 15 2 4 4 5
- 中略 -
cAD150 4 9 4 2 11
$pooled.cov #エヴェリット (2007) の共分散構造:等分散性
MB BH BL NH
MB 21.110805 0.036782 0.07908 2.0090
BH 0.036782 23.484598 5.20000 2.8451
BL 0.079080 5.200000 24.17908 1.1333
NH 2.008966 2.845057 1.13333 10.1526
その他
ans <- predict(reslda) #lda関数による判別の結果を返す
candis関数では以下に一致
$p.Bayes はベイズ確率。predict(reslda)の$posterior。判別後のケースごとの確率
$can.score は判別得点。predict(reslda)の$xと一致
$classificationは分類。predict(reslda)の$class (だと思う)
# groupは元の群、classificationは判別関数による分類
candis関数のprintについて
printで判別結果がうまくでないときがある。resultオブジェクト生成の問題のようだ
(正準判別分析をやってみた1)
データはエヴェリット (2007) の頭蓋骨データ
青木先生のcandis関数
rescandis <- candis(skulls[2:5],skulls[1])
# 結果print(rescandis)
基本はhttp://aoki2.si.gunma-u.ac.jp/R/candis.htmlを参照
lda関数、エヴェリット (2007) と関連するところだけ列挙
判別係数 #ldaのCoefficients of linear discriminants、エヴェリット (2007) の正準係数
axis 1 axis 2 axis 3 axis 4
MB -0.126676 0.038738 0.092768 -0.14883986
- 中略 -
NH -0.082851 -0.077293 -0.294589 -0.06685888
constant 2.283887 -22.530493 4.409749 36.07126158
判別結果 #エヴェリット (2007) のプラグイン推定による分類表
classification
group
c1850BC c200BC c3300BC c4000BC cAD150
c1850BC 15 2 4 4 5
- 中略 -
cAD150 4 9 4 2 11
$pooled.cov #エヴェリット (2007) の共分散構造:等分散性
MB BH BL NH
MB 21.110805 0.036782 0.07908 2.0090
BH 0.036782 23.484598 5.20000 2.8451
BL 0.079080 5.200000 24.17908 1.1333
NH 2.008966 2.845057 1.13333 10.1526
その他
ans <- predict(reslda) #lda関数による判別の結果を返す
candis関数では以下に一致
$p.Bayes はベイズ確率。predict(reslda)の$posterior。判別後のケースごとの確率
$can.score は判別得点。predict(reslda)の$xと一致
$classificationは分類。predict(reslda)の$class (だと思う)
# groupは元の群、classificationは判別関数による分類
candis関数のprintについて
printで判別結果がうまくでないときがある。resultオブジェクト生成の問題のようだ
正準判別分析をやってみた
データは
B.エヴェリット (2007) のp108-110 表5.8 頭蓋骨データ
リンク中のchap5skulls
従属変数になる群データはEPOCH (1列目) 、これをMB, BH, BL, NH (2-5列目) という連続変量で予測する
MASSパッケージのlda関数
# 線型判別の関数だが、3群以上あると正準判別をやってくれるらしい
reslda <- lda(skulls[,2:5], skulls[,1])
# これだけ?
print(reslda) # 結果
Call: lda(skulls[, 2:5], skulls[, 1])
# 事前確率
Prior probabilities of groups:
c4000BC c3300BC c1850BC c200BC cAD150
0.2 0.2 0.2 0.2 0.2
# 群内の平均値
Group means:
MB BH BL NH
c4000BC 131.3667 133.6000 99.16667 50.53333
-中略-
cAD150 136.1667 130.3333 93.50000 51.36667
# 判別係数 (正準係数とか正準判別係数とも)
Coefficients of linear discriminants:
LD1 LD2 LD3 LD4
MB 0.12667629 0.03873784 0.09276835 0.1488398644
-中略- BH, BLの結果
NH 0.08285128 -0.07729281 -0.29458931 0.0668588797
Proportion of trace:
LD1 LD2 LD3 LD4
0.8823 0.0809 0.0326 0.0042
結果がうまくhtmlででないなぁ…
データは
B.エヴェリット (2007) のp108-110 表5.8 頭蓋骨データ
リンク中のchap5skulls
従属変数になる群データはEPOCH (1列目) 、これをMB, BH, BL, NH (2-5列目) という連続変量で予測する
MASSパッケージのlda関数
# 線型判別の関数だが、3群以上あると正準判別をやってくれるらしい
reslda <- lda(skulls[,2:5], skulls[,1])
# これだけ?
print(reslda) # 結果
Call: lda(skulls[, 2:5], skulls[, 1])
# 事前確率
Prior probabilities of groups:
c4000BC c3300BC c1850BC c200BC cAD150
0.2 0.2 0.2 0.2 0.2
# 群内の平均値
Group means:
MB BH BL NH
c4000BC 131.3667 133.6000 99.16667 50.53333
-中略-
cAD150 136.1667 130.3333 93.50000 51.36667
# 判別係数 (正準係数とか正準判別係数とも)
Coefficients of linear discriminants:
LD1 LD2 LD3 LD4
MB 0.12667629 0.03873784 0.09276835 0.1488398644
-中略- BH, BLの結果
NH 0.08285128 -0.07729281 -0.29458931 0.0668588797
Proportion of trace:
LD1 LD2 LD3 LD4
0.8823 0.0809 0.0326 0.0042
結果がうまくhtmlででないなぁ…
Rjipwiki: Rで共分散構造分析・構造方程式モデル
psychパッケージのガイド (pdf)
semパッケージのマニュアル
J. Fox (2006), Structural Equation Modeling With the sem Package in R. Structural Equation Modeling, 13:465-486.
Quick-Rの因子分析のページ
James H. Steigerの確認的因子分析ガイド (from statpower)
psychパッケージのガイド (pdf)
semパッケージのマニュアル
J. Fox (2006), Structural Equation Modeling With the sem Package in R. Structural Equation Modeling, 13:465-486.
Quick-Rの因子分析のページ
James H. Steigerの確認的因子分析ガイド (from statpower)
最尤解はfactanalを使う。
result <- factanal(df, factors=2, method="mle", rotation="varimax")
# 2因子指定、最尤法、バリマックス回転
主因子法と重みつき最小二乗法はpsychパッケージのfa関数を使う
#主因子法。fa関数でfm="pa"としたもの
factor.pa(df, nfactors=因子数, rotation="回転法")
#重みつき最小二乗法。上と同様
factor.wls(data, nfactors=因子数, rotation="回転法")
以下、psychパッケージのヘルプより。わからないとこは?
r ローデータか相関行列
nfactors 因子数指定
residual 残差行列を表示するかどうか
rotate 回転。ダブルクォーテーションつきで指定する。none, varimax, quartimax, bentlerT, geominTが直交。promax, oblimin, simplimax, bentlerQ, geominQ, clusterが斜交
n.obs 相関行列を指定した場合は観測値の数を指定する
scores 因子得点
SMC 1にすると共通性の初期値を指定して固有値を報告する。TRUEのときはsquared multiple correlations
digits 小数点桁数指定
max.iter 反復回数の最大値
symmetric 対称性
warning 因子数が多すぎると警告を出してくれるらしい
fm 推定法の指定。重みつき最小二乗法はwls, glsは重みなし。paは主因子, minresはOLS
参考URL
nfactorsというライブラリを使うと固有値を出してスクリープロットがかけそう
http://www.statmethods.net/advstats/factor.html
result <- factanal(df, factors=2, method="mle", rotation="varimax")
# 2因子指定、最尤法、バリマックス回転
主因子法と重みつき最小二乗法はpsychパッケージのfa関数を使う
#主因子法。fa関数でfm="pa"としたもの
factor.pa(df, nfactors=因子数, rotation="回転法")
#重みつき最小二乗法。上と同様
factor.wls(data, nfactors=因子数, rotation="回転法")
以下、psychパッケージのヘルプより。わからないとこは?
r ローデータか相関行列
nfactors 因子数指定
residual 残差行列を表示するかどうか
rotate 回転。ダブルクォーテーションつきで指定する。none, varimax, quartimax, bentlerT, geominTが直交。promax, oblimin, simplimax, bentlerQ, geominQ, clusterが斜交
n.obs 相関行列を指定した場合は観測値の数を指定する
scores 因子得点
SMC 1にすると共通性の初期値を指定して固有値を報告する。TRUEのときはsquared multiple correlations
digits 小数点桁数指定
max.iter 反復回数の最大値
symmetric 対称性
warning 因子数が多すぎると警告を出してくれるらしい
fm 推定法の指定。重みつき最小二乗法はwls, glsは重みなし。paは主因子, minresはOLS
参考URL
nfactorsというライブラリを使うと固有値を出してスクリープロットがかけそう
http://www.statmethods.net/advstats/factor.html