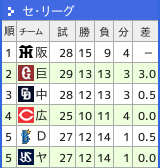
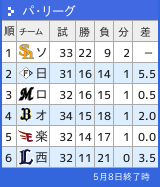
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
自分でたいしたものでもない関数をつくった
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1252696384")
# クリップボードからデータを読む。dat <- read.table("clipboard", header=T) と同じ。psychパッケージのread.clipboardの名前を変えただけ
dat <- read.c()
# クリップボードにタブ区切りでデータフレームを書き出す。write.table(dat, "clipboard", sep="\t", row.names=FALSE, quote=FALSE) と同じ
write.c(dat)
# クリップボードにオブジェクトを書き出す。sink("clipboard", split=TRUE) と同じ
# 使った後は必ずsink()とする。
sink.c(obj)
# 変数ごとに度数分布を書く
frq
# frq(1:5, iris)
大本は青木先生のfrequency関数である。LatexのデフォルトをFALSEにしただけ。感謝。
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1252696384")
# クリップボードからデータを読む。dat <- read.table("clipboard", header=T) と同じ。psychパッケージのread.clipboardの名前を変えただけ
dat <- read.c()
# クリップボードにタブ区切りでデータフレームを書き出す。write.table(dat, "clipboard", sep="\t", row.names=FALSE, quote=FALSE) と同じ
write.c(dat)
# クリップボードにオブジェクトを書き出す。sink("clipboard", split=TRUE) と同じ
# 使った後は必ずsink()とする。
sink.c(obj)
# 変数ごとに度数分布を書く
frq
# frq(1:5, iris)
大本は青木先生のfrequency関数である。LatexのデフォルトをFALSEにしただけ。感謝。
PR
Jonathan Baronの分散分析の解説でSPSSで平方和をタイプ2と指定すると
aov関数での結果と同じになる、と書いてあり、疑問に思ったがよくわからなかったので
Rjpwiki と 青木先生の掲示板で聞いてみた。感謝。
周辺度数が比例しているとき ("要因"が直交していると) 、タイプ1とタイプ2は同じになるようだ。
Jonathan Baronのもよく読んでみると
平方和の疑問が解決したので、ずっと下書き状態だった混合3要因分散分析のコードを書いておこう。 基本はJonathan Baronのもの。少し原典のコードの誤りを修正した。
#データ生成
data1<-c(49,47,46,47,48,47,41,46,43,47,46,45,48,46,47,45,49,44,44,45,42,45,45,40,49,46,47,45,49,45,41,43,44,46,45,40,45,43,44,45,48,46,40,45,40,45,47,40)
## 見やすくした (上記HPのコードを少し修正した。HPのままこぴぺするとエラーになる)
matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2")))
# aov用のデータフレーム作成
# 水準も従属変数も全て縦に並べる
Hays.df <- data.frame(rt = data1, subj = factor(rep(paste("subj", 1:12, sep=""), 4)), shape = factor(rep(rep(c("shape1", "shape2"), c(12, 12)), 2)), color = factor(rep(c("color1", "color2"), c(24, 24))))
# 非釣合型のデータにするため、grp変数を付け加える
Hays.df$grp <- factor(rep(c(1,1,1,1,1,1,1,1,2,2,2,2), 4))
# aovで分析
aovres <- aov(rt ~ grp*color*shape + Error(subj/(color+shape)), data=Hays.df)
summary(aovres)
## HPの記述は誤差項のカッコをつけわすれている; summary(aov(rt ~ grp*color*shape + Error(subj/shape+color), data=Hays.df))
aov関数での結果と同じになる、と書いてあり、疑問に思ったがよくわからなかったので
Rjpwiki と 青木先生の掲示板で聞いてみた。感謝。
周辺度数が比例しているとき ("要因"が直交していると) 、タイプ1とタイプ2は同じになるようだ。
Jonathan Baronのもよく読んでみると
The example aov() analysis below can be compared with the results of SPSS using SSTYPE(2)
「以下のaovでの分析例」では…と言っている。私の無知である。ジョナサンッッ!すまないッッ!!
でもその前にSPSS produces the same output as R if the user tells SPSS to calculate the Type I SS (SSTYPE(1)) or Type II SS (SSTYPE(2)) instead of the default SSTYPE(3).って言ってるよね…
平方和の疑問が解決したので、ずっと下書き状態だった混合3要因分散分析のコードを書いておこう。 基本はJonathan Baronのもの。少し原典のコードの誤りを修正した。
#データ生成
data1<-c(49,47,46,47,48,47,41,46,43,47,46,45,48,46,47,45,49,44,44,45,42,45,45,40,49,46,47,45,49,45,41,43,44,46,45,40,45,43,44,45,48,46,40,45,40,45,47,40)
## 見やすくした (上記HPのコードを少し修正した。HPのままこぴぺするとエラーになる)
matrix(data1, ncol= 4, dimnames = list(paste("subj", 1:12), c("Shape1.Color1", "Shape2.Color1" ,"Shape1.Color2", "Shape2.Color2")))
# aov用のデータフレーム作成
# 水準も従属変数も全て縦に並べる
Hays.df <- data.frame(rt = data1, subj = factor(rep(paste("subj", 1:12, sep=""), 4)), shape = factor(rep(rep(c("shape1", "shape2"), c(12, 12)), 2)), color = factor(rep(c("color1", "color2"), c(24, 24))))
# 非釣合型のデータにするため、grp変数を付け加える
Hays.df$grp <- factor(rep(c(1,1,1,1,1,1,1,1,2,2,2,2), 4))
# aovで分析
aovres <- aov(rt ~ grp*color*shape + Error(subj/(color+shape)), data=Hays.df)
summary(aovres)
## HPの記述は誤差項のカッコをつけわすれている; summary(aov(rt ~ grp*color*shape + Error(subj/shape+color), data=Hays.df))
dat <- read.table("http://personality-project.org/R/datasets/R.appendix1.data", header=T)
colnames(dat) <- c("x", "y") # 列名を短くする
aovres <- aov(y ~ x, dat)
summary(aovres)
pairwise.t.test(dat$y, dat$x, p.adj = "holm")
# p値だけで自由度もt値も出ないとはどういうことだ
以下参考
http://www.personality-project.org/r/#anova
colnames(dat) <- c("x", "y") # 列名を短くする
aovres <- aov(y ~ x, dat)
summary(aovres)
pairwise.t.test(dat$y, dat$x, p.adj = "holm")
# p値だけで自由度もt値も出ないとはどういうことだ
以下参考
http://www.personality-project.org/r/#anova
最近全然やらないので書き忘れていたが、t検定をメモしておこう
対応のない場合 (独立2標本) のときと対応のあるときで記述が違うので注意
等分散を仮定するときはvar.equal=TRUEとする。しないけど
対応のない場合
t.test(y ~ x, dat)
## xが独立変数、yが従属変数。xは因子変数にしなくてもよい
対応のある場合
t.test(datp[,1], datp[,2], paired=TRUE)
## ここで、pairedを指定しないと対応なしの検定になり、同じ結果を返す
## pairedの前で片側検定を指定できる (デフォルトは両側) 。まあ両側やっときゃいいだろう
## "greater"は前者が後者より大きい (datp[,1]>[,2]) 、"less"だとその逆。y~xの形式でどう指定するかは不明。というかできない (たぶん)
## データフレームは以下のような感じ
# このブログのエディタはエクセルから貼り付けたら勝手にtableつくってくれた。便利
対応のない場合 (独立2標本) のときと対応のあるときで記述が違うので注意
等分散を仮定するときはvar.equal=TRUEとする。しないけど
対応のない場合
t.test(y ~ x, dat)
## xが独立変数、yが従属変数。xは因子変数にしなくてもよい
対応のある場合
t.test(datp[,1], datp[,2], paired=TRUE)
## ここで、pairedを指定しないと対応なしの検定になり、同じ結果を返す
## pairedの前で片側検定を指定できる (デフォルトは両側) 。まあ両側やっときゃいいだろう
## "greater"は前者が後者より大きい (datp[,1]>[,2]) 、"less"だとその逆。
通りすがりさん、ありがとうございます
t.test(y~x, dat, alternative="greater")
ところで、グループ1 > グループ2 を検定しているが、
xのカテゴリーに割り当てた数値 (e.g., 0と1) のどちらがグループ1かわからない (たぶん数値の若い方をグループ1としてるんだと思うが)
# これで調べよう
aggregate(dat[,2], list(dat[,1]), mean, na.rm=TRUE) # 平均
t.test(y~x, dat, alternative="greater")
ところで、グループ1 > グループ2 を検定しているが、
xのカテゴリーに割り当てた数値 (e.g., 0と1) のどちらがグループ1かわからない (たぶん数値の若い方をグループ1としてるんだと思うが)
# これで調べよう
aggregate(dat[,2], list(dat[,1]), mean, na.rm=TRUE) # 平均
## データフレームは以下のような感じ
| 対応なし dat |
対応あり datp |
|||
| x | y | y1 | y2 | |
| 1 | 7 | 7 | 3 | |
| 1 | 5 | 5 | 2 | |
| 1 | 5 | 5 | 1 | |
| 1 | 9 | 9 | 4 | |
| 1 | 5 | 5 | 1 | |
| 0 | 3 | |||
| 0 | 2 | |||
| 0 | 1 | |||
| 0 | 4 | |||
| 0 | 1 | |||
# このブログのエディタはエクセルから貼り付けたら勝手にtableつくってくれた。便利
標準誤差の関数を見つけたのでメモしておく
# 関数
se <- function(x)
{
y <- x[!is.na(x)] # remove the missing values
sqrt(var(as.vector(y))/length(y))
}
# sapplyにした
sapply(dat, function(x) sqrt(var(as.vector(na.omit(x)))/length(na.omit(x))))
こっちも変えた
http://scratchhit.pazru.com/Entry/20/
# 関数
se <- function(x)
{
y <- x[!is.na(x)] # remove the missing values
sqrt(var(as.vector(y))/length(y))
}
# sapplyにした
sapply(dat, function(x) sqrt(var(as.vector(na.omit(x)))/length(na.omit(x))))
こっちも変えた
http://scratchhit.pazru.com/Entry/20/