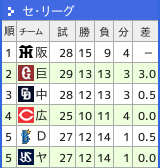
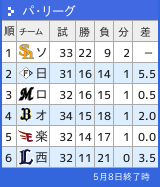
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
par(mfcol=c(3,3))
plot(1:10, type="p", main="p") # points
plot(1:10, type="l", main="l") # lines
plot(1:10, type="b", main="b") # both. pointsとline
plot(1:10, type="c", main="c") # the lines part alone of "b"
plot(1:10, type="o", main="o") # overplotted. 線とポイントを重ねる
plot(1:10, type="h", main="h") # histogram
plot(1:10, type="s", main="s") # stair steps
plot(1:10, type="S", main="S") # other steps
plot(1:10, type="n", main="n") # no plotting
plot(1:10, type="p", main="p") # points
plot(1:10, type="l", main="l") # lines
plot(1:10, type="b", main="b") # both. pointsとline
plot(1:10, type="c", main="c") # the lines part alone of "b"
plot(1:10, type="o", main="o") # overplotted. 線とポイントを重ねる
plot(1:10, type="h", main="h") # histogram
plot(1:10, type="s", main="s") # stair steps
plot(1:10, type="S", main="S") # other steps
plot(1:10, type="n", main="n") # no plotting
PR
基本はもちろんRjpwikiのカラーチャートのページ。感謝
# 虹色はrainbow
barplot(1:7, col=rainbow(7))
# これで選べるのは
# rainbow, heat.colors, terrain.colors, topo.colors, cm.colors, grey.colors
# たぶんrainbow, grey, cm.colorsくらいしか使わないだろう
barplot(1:7, col=cm.colors(7))
barplot(1:7, col=grey.colors(7))
RColorBrewerというのがあるらしい
Rjpwikiの紹介ページ
CRANのページ
大本はColorBrewerというwebツールみたい
library(RColorBrewer)
example(brewer.pal)
# 要は、Rのデフォルトの色配分よりコントラストが高く視認性がいいということらしい
brewer.pal関数で色の指定ベクトルを返す
brewer.pal(n=3, name="Greens")
# nameで色を指定する。パターンは以下のコマンドで一覧を確認できる
display.brewer.all(type="all")
# BuPu, BuGn, Blues, Greys, Spectralあたりがええかのう
par(mfcol=c(3,3))
display.brewer.pal(7, "BuPu")
display.brewer.pal(7, "BuGn")
display.brewer.pal(7, "Blues")
display.brewer.pal(7, "Greys")
display.brewer.pal(7, "Pastel1")
display.brewer.pal(7, "Pastel2")
display.brewer.pal(7, "Spectral")
display.brewer.pal(7, "YlGnBu")
# デフォルトのgrey.colorsと比較してみる
par(mfcol=c(3,2))
barplot(1:3, main="rcb_grey", col=brewer.pal(3, "Greys"))
barplot(1:6, col=brewer.pal(6, "Greys"))
barplot(1:9, col=brewer.pal(9, "Greys"))
barplot(1:3, main="grey.colors", col=rev(grey.colors(3)))
barplot(1:6, col=rev(grey.colors(6)))
barplot(1:9, col=rev(grey.colors(9)))
## rev() はベクトルを逆に並べ替える関数。知らなかった
# 虹色はrainbow
barplot(1:7, col=rainbow(7))
# これで選べるのは
# rainbow, heat.colors, terrain.colors, topo.colors, cm.colors, grey.colors
# たぶんrainbow, grey, cm.colorsくらいしか使わないだろう
barplot(1:7, col=cm.colors(7))
barplot(1:7, col=grey.colors(7))
RColorBrewerというのがあるらしい
Rjpwikiの紹介ページ
CRANのページ
大本はColorBrewerというwebツールみたい
library(RColorBrewer)
example(brewer.pal)
# 要は、Rのデフォルトの色配分よりコントラストが高く視認性がいいということらしい
brewer.pal関数で色の指定ベクトルを返す
brewer.pal(n=3, name="Greens")
# nameで色を指定する。パターンは以下のコマンドで一覧を確認できる
display.brewer.all(type="all")
# BuPu, BuGn, Blues, Greys, Spectralあたりがええかのう
par(mfcol=c(3,3))
display.brewer.pal(7, "BuPu")
display.brewer.pal(7, "BuGn")
display.brewer.pal(7, "Blues")
display.brewer.pal(7, "Greys")
display.brewer.pal(7, "Pastel1")
display.brewer.pal(7, "Pastel2")
display.brewer.pal(7, "Spectral")
display.brewer.pal(7, "YlGnBu")
# デフォルトのgrey.colorsと比較してみる
par(mfcol=c(3,2))
barplot(1:3, main="rcb_grey", col=brewer.pal(3, "Greys"))
barplot(1:6, col=brewer.pal(6, "Greys"))
barplot(1:9, col=brewer.pal(9, "Greys"))
barplot(1:3, main="grey.colors", col=rev(grey.colors(3)))
barplot(1:6, col=rev(grey.colors(6)))
barplot(1:9, col=rev(grey.colors(9)))
## rev() はベクトルを逆に並べ替える関数。知らなかった
くっつけたい文字の次に "2 p " (2半角スペースp半角スペース) と入力
2からpの後の半角スペースまで選択し
ホームタブ、段落リボンの拡張書式ボタン (幅広A) の下向き矢印をクリックし
組み文字 を選択、OK
アルファ、ベータ、イータなどを入力する場合は、
記号と特殊文字から「ギリシャおよびコプト」を選択
2からpの後の半角スペースまで選択し
ホームタブ、段落リボンの拡張書式ボタン (幅広A) の下向き矢印をクリックし
組み文字 を選択、OK
アルファ、ベータ、イータなどを入力する場合は、
記号と特殊文字から「ギリシャおよびコプト」を選択
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669") source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1273264153") ## データはテクニカルブック p117より。感謝 dat <- data.frame( a1b1=c(3, 3, 1, 3, 5), a1b2=c(4, 3, 4, 5, 7), a1b3=c(6, 6, 6, 4, 8), a1b4=c(5, 7, 8, 7, 9), a2b1=c(3, 5, 2, 4, 6), a2b2=c(2, 6, 3, 6, 4), a2b3=c(3, 2, 3, 6, 5), a2b4=c(2, 3, 3, 4, 6) ) dat (mmat <- matrix(mean(dat), nr=4, dimnames=list(bnms, anms))) (sdmat <- matrix(sd(dat), nr=4, dimnames=list(bnms, anms))) ## とりあえずプロット barplot(mmat, beside=T) dev.off() ## 水準とかの情報 lvnms <- c("a1b1", "a1b2", "a1b3", "a1b4", "a2b1", "a2b2", "a2b3", "a2b4") ncl <- ncol(dat) nlva <- 2 # 自分で入力 nlvb <- 4 # 自分で入力 anms <- c("a1", "a2") # 自分で入力 bnms <- c("b1", "b2", "b3", "b4") # 自分で入力 ## データフレームの分割 clnm <- 1:ncl clmt <- matrix(clnm, nlvb) adatlist <- list() bdatlist <- list() for (i in 1:nlva) { x <- dat[clmt[,i]] ; names(x) <- bnms adatlist[[i]] <- x } names(adatlist) <- anms for (i in 1:nlvb) { x <- dat[clmt[i,]] ; names(x) <- anms bdatlist[[i]] <- x } names(bdatlist) <- bnms adatlist bdatlist ## 全体の分散分析 lmres <- lm(as.matrix(dat[lvnms])~1, dat) library(car) afact <- factor(rep(anms, each=4)) bfact <- factor(rep(bnms, 2)) idat <- data.frame(afact, bfact) Anovares <- Anova(lmres, idata=idat, idesign=~afact*bfact) Ares <- Astat(Anovares) Aunv <- Ares$unv Aunv ## 要因の主効果の多重比較: 水準別誤差項 # a要因 tmp <- list() for (nm in anms) { tmp[[nm]] <- rowMeans(adatlist[[nm]]) } ameans <- data.frame(tmp) factmc.a <- holm.mc(ameans, datw=T, psd=F, paired=T) factmc.a # b要因 tmp <- list() for (nm in bnms) { tmp[[nm]] <- rowMeans(bdatlist[[nm]]) } bmeans <- data.frame(tmp) factmc.b <- holm.mc(bmeans, datw=T, psd=F, paired=T) factmc.b ## 単純主効果 ## ## 水準別誤差項で単純主効果 ## ## bにおけるaの効果とその多重比較 afct <- factor(anms); iadat <- data.frame(afct) seffect.alist <- list(); seffect.a <- list() seffect.alist <- lapply(bdatlist, function(x) Anova(lm(as.matrix(x)~1, x), idata=iadat, idesign=~afct)) mcse.a <- list() mcse.a <- lapply(bdatlist, function(x) holm.mc(x, datw=T, psd=F, paired=T)) seffect.a <- lapply(seffect.alist, function(x) Astat(x)$unv) seffect.a # 単純主効果 mcse.a # 多重比較 ## aにおけるbの効果とその多重比較 bfct <- factor(bnms); ibdat <- data.frame(bfct) seffect.blist <- list(); seffect.b <- list() seffect.blist <- lapply(adatlist, function(x) Anova(lm(as.matrix(x)~1, x), idata=ibdat, idesign=~bfct)) mcse.b <- list() mcse.b <- lapply(adatlist, function(x) holm.mc(x, datw=T, psd=F, paired=T)) seffect.b <- lapply(seffect.blist, function(x) Astat(x)$unv) seffect.b # 単純主効果 mcse.b # 多重比較 ## 交互作用対比 ### 処理用オブジェクト icreslist <- list(); n <- 1 lisnm <- vector() cmba <- combn(anms, 2) cmbb <- combn(bnms, 2) ### 交互作用対比の分析 for (i in 1:ncol(cmba)) { x1 <- adatlist[[cmba[1,i]]] x2 <- adatlist[[cmba[2,i]]] for (j in 1:ncol(cmbb)) { y1 <- x1[cmbb[,j]] y2 <- x2[cmbb[,j]] dattmp <- data.frame(y1, y2) names(dattmp) <- c(paste(cmba[1,i], cmbb[,j], sep=""), paste(cmba[2,i], cmbb[,j], sep="")) afctic <- factor(rep(cmba[,i], each=2)) bfctic <- factor(rep(cmbb[,j], 2)) idatic <- data.frame(afctic, bfctic) Anovaresic <- Anova(lm(as.matrix(dattmp)~1, dattmp), idata=idatic, idesign=~afctic*bfctic) Aresic <- Astat(Anovaresic)$unv lisnm <- paste(paste(paste(cmba[1,i], cmbb[,j], sep=""), collapse="_"), "_vs._",paste(paste(cmba[2,i], cmbb[,j], sep=""), collapse="_"), sep="") icreslist[[lisnm]] <- Aresic; n <- n+1 } } #names(icreslist) <- lisnm icreslist tmp <- lapply(icreslist, function(x) x[4,]) tmp2 <- t(data.frame(tmp)) pvholm <- p.adjust(tmp2[,6], "holm") icres <- cbind(tmp2, pvholm) colnames(icres) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") class(icres) <- "anova" icres ## プールされた誤差項で分析してみる。多重比較はやめておこう Ares Aunv ### それぞれの誤差項をとりだしておく (mse.a <- (Aunv[2,3]+Aunv[4,3])/(Aunv[2,4]+Aunv[4,4])) (mse.adf <- (Aunv[2,4]+Aunv[4,4])) (mse.b <- (Aunv[3,3]+Aunv[4,3])/(Aunv[3,4]+Aunv[4,4])) (mse.bdf <- (Aunv[3,4]+Aunv[4,4])) (mse.ab <- (Aunv[4,3])/(Aunv[4,4])) (mse.abdf <- (Aunv[4,4])) ## プールされた誤差項で単純主効果 ## ### 水準別誤差項の結果を利用する # bにおけるAの単純効果 tmp <- list() tmp <- lapply(seffect.a, function(x) Astat(x)$unv[2,]) tmp2 <- t(data.frame(tmp)) pvholm <- p.adjust(1-pf((tmp2[,1]/tmp2[,2])/mse.a, tmp2[,2], mse.adf), "holm") seffect.a.pl <- cbind(tmp2[,1:2], mse.a*mse.adf, mse.adf, mse.a, (tmp2[,1]/tmp2[,2])/mse.a, 1-pf((tmp2[,1]/tmp2[,2])/mse.a, tmp2[,2], mse.adf), pvholm) colnames(seffect.a.pl) <- c("ss", "nmdf", "errorss", "dendf", "mse", "fv", "pv", "Pr(>F).holm") rownames(seffect.a.pl) <- paste("a effect at ", bnms, sep="") class(seffect.a.pl) <- "anova" seffect.a.pl # Bの効果 at a水準 tmp <- list() tmp <- lapply(seffect.b, function(x) Astat(x)$unv[2,]) tmp2 <- t(data.frame(tmp)) pvholm <- p.adjust(1-pf((tmp2[,1]/tmp2[,2])/mse.b, tmp2[,2], mse.bdf), "holm") seffect.b.pl <- cbind(tmp2[,1:2], mse.b*mse.bdf, mse.bdf, mse.b, (tmp2[,1]/tmp2[,2])/mse.b, 1-pf((tmp2[,1]/tmp2[,2])/mse.b, tmp2[,2], mse.bdf), pvholm) colnames(seffect.b.pl) <- c("ss", "nmdf", "errorss", "dendf", "mse", "fv", "pv", "Pr(>F).holm") rownames(seffect.b.pl) <- paste("b effect at ", anms, sep="") class(seffect.b.pl) <- "anova" seffect.b.pl ## プールされた誤差項で交互作用対比…交互作用項の誤差項を使えばいいのか? icres.pltp <- icres[,1:2] pl.mss <- icres[,1]/icres[,2] pl.errorss <- mse.ab*mse.abdf pv.holm <- p.adjust(1-(pf(pl.mss/mse.ab, icres[,2], mse.abdf)), "holm") icres.pl <- cbind(icres.pltp, pl.errorss, mse.abdf, pl.mss/mse.ab, 1-(pf(pl.mss/mse.ab, icres[,2], mse.abdf)), pv.holm) colnames(icres.pl) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") class(icres.pl) <- "anova" icres.pl
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669") source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1273264153") ## データ ## 参考: http://www.psychology.emory.edu/clinical/mcdowell/PSYCH560/mixed.htm 感謝。 dat <- data.frame( cnd = factor(rep(c("wt", "atp", "trt"), each=4)), pre=c(13, 10, 13, 4, 5, 8, 14, 12, 13, 9, 14, 8), hlf=c(14, 11, 19, 12, 10, 15, 16, 21, 24, 22, 22, 18), pst=c(20, 14, 21, 15, 21, 24, 23, 26, 30, 24, 28, 28), fu=c(17, 15, 18, 14, 17, 22, 23, 26, 28, 22, 28, 27) ) by(dat[,2:5], list(dat[,1]), mean) by(dat[,2:5], list(dat[,1]), sd) ## とりあえずプロット x <-aggregate(dat[2:5], list(dat[,1]), mean) gx <- as.matrix(x[2:5]) rownames(gx) <- names(dat[,1]) barplot(gx, beside=T) dev.off() ## 水準名とか ncl <- ncol(dat) anms <- levels(dat[,1]) nanms <- sapply(anms, function(x) sum(x==dat[,1])) cmba <- combn(anms, 2) bnms <- names(dat[2:ncl]) mmbs <- mean(aggregate(dat[,2:ncl], list(dat[,1]), mean)[,2:ncl]) cmbb <- combn(bnms, 2) ## 全体の分散分析 options(contrasts = c("contr.sum", "contr.sum")) lmres <- lm(as.matrix(dat[,2:ncl]) ~ dat[,1] , data = dat) bfact <- factor(bnms) idat <- data.frame(bfact) library(car) Anovares <- Anova(lmres, idata = idat, idesign = ~ bfact) (Ares <- Astat(Anovares)$unv) ## 要因の主効果の多重比較: 参加者間要因 val <- rowMeans(dat[,2:ncl]) a <- dat[,1] factmc.a <- holm.mc(data.frame(a, val)) factmc.a ## 要因の主効果の多重比較: 参加者内要因 factmc.blist <- list() ps <- vector() library(car) for (i in 1:ncol(cmbb)) { mcbfact <- factor(cmbb[,i]) mcidata <- data.frame(mcbfact) mclmres <- lm(as.matrix(dat[,c(cmbb[,i])])~dat[,1], dat) mcAnovares <- Anova(mclmres, idata=mcidata, idesign=~mcbfact) mcAres <- Astat(mcAnovares)$unv mcmse <- mcAres[2,3]/mcAres[2,4] mcdf <- mcAres[2,4] den1 <- (mean(1/nanms)/nlevels(dat[,1]))*2 diff <- mmbs[cmbb[1,i]] - mmbs[cmbb[2,i]] tv <- diff/sqrt(mcmse*den1) ps[i] <- pt(abs(tv), mcdf, lower.tail = F)*2 resv <- c(mmbs[cmbb[1,i]], mmbs[cmbb[2,i]], diff, mcmse, mcdf, tv, pt(abs(tv), mcdf, lower.tail = F)*2) factmc.blist [[i]] <- resv } factmc.b <- t(data.frame(factmc.blist )) ps.holm <- p.adjust(ps, "holm") factmc.b <- cbind(factmc.b, ps.holm) colnames(factmc.b) <- c("m1", "m2", "diff", "mse", "df", "tv", "pv", "Pr(>|t|).holm") rownames(factmc.b) <- paste(cmbb[1,], "_", cmbb[2,], sep="") class(factmc.b) <- "anova" factmc.b ## 単純主効果 ## ## 水準別誤差項で単純主効果 ## ## b (参加者内水準) におけるaの効果とその多重比較 seffect.a <- list() seffect.a <- lapply(dat[,2:ncl], function(x) Anova(lm(x~a, dat))) mcse.a <- list() for (nm in bnms) { val <- dat[nm] x <- dat[,1] mcse.a[[nm]] <- holm.mc(data.frame(x, val)) } names(seffect.a) <- paste(names(dat)[1], " effect at ", bnms, sep="") names(mcse.a) <- paste(names(dat)[1], " effect at ", bnms, sep="") seffect.a # 単純主効果 mcse.a # その多重比較 ## a (参加者間水準) におけるBの効果とその多重比較 seffect.b <- list(); seffect.b <- list() mcse.b <- list() for (nm in anms) { dattmp <- subset(dat, dat[,1]==nm) Anovares.sepa <- Anova(lm(as.matrix(dattmp[,2:ncl])~1, data=dattmp), idata=idat, idesign=~bfact) seffect.b[[nm]] <- Anovares.sepa mcse.b[[nm]] <- holm.mc(dattmp[,2:ncl], datw=T, psd=F, paired=T) } seffect.b <- lapply(seffect.b, function(x) Astat(x)$unv) names(seffect.b) <- paste(names(dat)[2], " effect at ", anms, sep="") names(mcse.b) <- paste(names(dat)[2], " effect at ", anms, sep="") seffect.b # 単純主効果 mcse.b # 多重比較 ## 交互作用対比 ### 処理用オブジェクト iclist <- list() ps <- vector() rnms <- vector() cmba cmbb icn <- 1 ### 交互作用対比の分析 for (i in 1:ncol(cmba)) { dattmpa <- subset(dat, dat[,1]==cmba[1,i]|dat[,1]==cmba[2,i]) for (j in 1:ncol(cmbb)) { dattmp <- data.frame(dattmpa[1], dattmpa[cmbb[,j]]) bfacttmp <- factor(cmbb[,j]) idattmp <- data.frame(bfacttmp) lmrestmp <- lm(as.matrix(dattmp[,2:3])~dattmp[,1], dattmp) Anovarestmp <- Anova(lmrestmp, idata=idattmp, idesign=~bfacttmp) Arestmp <- Astat(Anovarestmp)$unv iclist[[icn]] <- Arestmp[3,]; ps[icn] <- Arestmp[3,6]; rnms[icn] <- paste(paste(cmba[,i], collapse=""), " vs. ", paste(cmbb[,j], collapse=""), sep="") icn <- icn+1 } } icres <- t(data.frame(iclist)) ps.holm <- p.adjust(ps, "holm") icres <- cbind(icres, ps) colnames(icres) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") rownames(icres) <- rnms class(icres) <- "anova" icres # それぞれのmseはicres[,3]/icres[,4] ## プールされた誤差項で分析してみる Ares ### それぞれの誤差項をとりだしておく mse.a <- Ares[1,3]/Ares[1,4] # 参加者間要因のmse: aの主効果の検定に使う df.a <- Ares[1,4] mse.ap <- sum(Ares[c(1,3), 3])/sum(Ares[c(1,3), 4]) # 参加者間要因を含むプールしたmse: aの単純主効果検定につかう df.ap <- sum(Ares[c(1,3), 4]) # mse.aの自由度 mse.b <- Ares[2,3]/Ares[2,4] # 参加者内要因のプールしたmse df.b <- Ares[2,4] # 参加者内要因の自由度 mse.ab <- Ares[3,3]/Ares[3,4] # 交互作用項のプールしたmse: bの単純主効果検定に使う df.ab <- Ares[3,4] # mse.abの自由度 ## 要因の主効果の多重比較: 参加者間要因 val <- rowMeans(dat[,2:ncl]) a <- dat[,1] factmc.a.pl <- holm.mc(data.frame(a, val), mse=mse.a, mse.df=df.a) factmc.a.pl ## 要因の主効果の多重比較: 参加者内要因 factmc.b.pllist <- list() ps <- vector() library(car) for (i in 1:ncol(cmbb)) { mcmse <- mse.ab mcdf <- df.ab den1 <- (mean(1/nanms)/nlevels(dat[,1]))*2 diff <- mmbs[cmbb[1,i]] - mmbs[cmbb[2,i]] tv <- diff/sqrt(mcmse*den1) ps[i] <- pt(abs(tv), mcdf, lower.tail = F)*2 resv <- c(mmbs[cmbb[1,i]], mmbs[cmbb[2,i]], diff, mcmse, mcdf, tv, pt(abs(tv), mcdf, lower.tail = F)*2) factmc.b.pllist [[i]] <- resv } factmc.b.pl <- t(data.frame(factmc.b.pllist )) ps.holm <- p.adjust(ps, "holm") factmc.b.pl <- cbind(factmc.b.pl, ps.holm) colnames(factmc.b.pl) <- c("m1", "m2", "diff", "mse", "df", "tv", "pv", "Pr(>|t|).holm") rownames(factmc.b.pl) <- paste(cmbb[1,], "_", cmbb[2,], sep="") class(factmc.b.pl) <- "anova" factmc.b.pl ## プールされた誤差項で単純主効果 ## ## b (参加者内水準) におけるAの単純効果 seffect.a.pllist <- list() for (nm in bnms) { Anovares.se <- Anova(lm(dat[,nm]~a, dat)) Ares.se <- Astat(Anovares.se)$unv seffect.a.pllist[[nm]] <- c(unlist(Ares.se[1,1:2]), mse.ap, df.ap, ((Ares.se[1,1]/Ares.se[1,2])/mse.ap), 1-pf(((Ares.se[1,1]/Ares.se[1,2])/mse.ap), Ares.se[1,2], df.ap)) } seffect.a.pl <-t(data.frame(seffect.a.pllist)) colnames(seffect.a.pl) <- c("ss", "nmdf", "mse", "dendf", "fv", "Pr(>F)") rownames(seffect.a.pl) <- paste(names(dat)[1], " effect at ", bnms, sep="") class(seffect.a.pl) <- "anova" seffect.a.pl ## 多重比較は勘弁してやろう ## a (参加者間水準) におけるBの単純効果 seffect.b.pllist <- list() for (nm in anms) { dattmp <- subset(dat, dat[,1]==nm) Anovares.se <- Anova(lm(as.matrix(dattmp [,2:ncl])~1, data=dattmp) , idata=idat, idesign=~bfact, type=2) Ares.se1 <- Astat(Anovares.se)$unv if (rownames(Ares.se1)[1]=="(Intercept)") {Ares.se <- Ares.se1[2:nrow(Ares.se1),]} else {Ares.se <- Ares.se1} seffect.b.pllist[[nm]] <- c(unlist(Ares.se[1:2]), mse.b, df.b, ((Ares.se[1]/Ares.se[2])/mse.b), 1-pf(((Ares.se[1]/Ares.se[2])/mse.b), Ares.se[2], df.b)) } seffect.b.pl <-t(data.frame(seffect.b.pllist)) colnames(seffect.b.pl ) <- c("ss", "nmdf", "mse", "dendf", "fv", "Pr(>F)") rownames(seffect.b.pl) <- paste(names(dat)[2], " effect at ", anms, sep="") class(seffect.b.pl ) <- "anova" seffect.b.pl ### プールされた誤差項での交互作用対比 ### 交互作用項のを使えばいいのだろうか? icres.pltp <- icres[,1:2] pl.mss <- icres[,1]/icres[,2] pl.errorss <- mse.ab*df.ab pv.holm <- p.adjust(1-(pf(pl.mss/mse.ab, icres[,2], df.ab)), "holm") icres.pl <- cbind(icres.pltp, pl.errorss, df.ab, pl.mss/mse.ab, 1-(pf(pl.mss/mse.ab, icres[,2], df.ab)), pv.holm) colnames(icres.pl) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") class(icres.pl) <- "anova" icres.pl