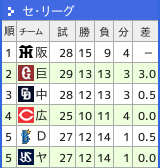
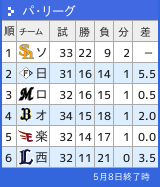
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669") source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1273264153") ## データはテクニカルブック p117より。感謝 dat <- data.frame( a1b1=c(3, 3, 1, 3, 5), a1b2=c(4, 3, 4, 5, 7), a1b3=c(6, 6, 6, 4, 8), a1b4=c(5, 7, 8, 7, 9), a2b1=c(3, 5, 2, 4, 6), a2b2=c(2, 6, 3, 6, 4), a2b3=c(3, 2, 3, 6, 5), a2b4=c(2, 3, 3, 4, 6) ) dat (mmat <- matrix(mean(dat), nr=4, dimnames=list(bnms, anms))) (sdmat <- matrix(sd(dat), nr=4, dimnames=list(bnms, anms))) ## とりあえずプロット barplot(mmat, beside=T) dev.off() ## 水準とかの情報 lvnms <- c("a1b1", "a1b2", "a1b3", "a1b4", "a2b1", "a2b2", "a2b3", "a2b4") ncl <- ncol(dat) nlva <- 2 # 自分で入力 nlvb <- 4 # 自分で入力 anms <- c("a1", "a2") # 自分で入力 bnms <- c("b1", "b2", "b3", "b4") # 自分で入力 ## データフレームの分割 clnm <- 1:ncl clmt <- matrix(clnm, nlvb) adatlist <- list() bdatlist <- list() for (i in 1:nlva) { x <- dat[clmt[,i]] ; names(x) <- bnms adatlist[[i]] <- x } names(adatlist) <- anms for (i in 1:nlvb) { x <- dat[clmt[i,]] ; names(x) <- anms bdatlist[[i]] <- x } names(bdatlist) <- bnms adatlist bdatlist ## 全体の分散分析 lmres <- lm(as.matrix(dat[lvnms])~1, dat) library(car) afact <- factor(rep(anms, each=4)) bfact <- factor(rep(bnms, 2)) idat <- data.frame(afact, bfact) Anovares <- Anova(lmres, idata=idat, idesign=~afact*bfact) Ares <- Astat(Anovares) Aunv <- Ares$unv Aunv ## 要因の主効果の多重比較: 水準別誤差項 # a要因 tmp <- list() for (nm in anms) { tmp[[nm]] <- rowMeans(adatlist[[nm]]) } ameans <- data.frame(tmp) factmc.a <- holm.mc(ameans, datw=T, psd=F, paired=T) factmc.a # b要因 tmp <- list() for (nm in bnms) { tmp[[nm]] <- rowMeans(bdatlist[[nm]]) } bmeans <- data.frame(tmp) factmc.b <- holm.mc(bmeans, datw=T, psd=F, paired=T) factmc.b ## 単純主効果 ## ## 水準別誤差項で単純主効果 ## ## bにおけるaの効果とその多重比較 afct <- factor(anms); iadat <- data.frame(afct) seffect.alist <- list(); seffect.a <- list() seffect.alist <- lapply(bdatlist, function(x) Anova(lm(as.matrix(x)~1, x), idata=iadat, idesign=~afct)) mcse.a <- list() mcse.a <- lapply(bdatlist, function(x) holm.mc(x, datw=T, psd=F, paired=T)) seffect.a <- lapply(seffect.alist, function(x) Astat(x)$unv) seffect.a # 単純主効果 mcse.a # 多重比較 ## aにおけるbの効果とその多重比較 bfct <- factor(bnms); ibdat <- data.frame(bfct) seffect.blist <- list(); seffect.b <- list() seffect.blist <- lapply(adatlist, function(x) Anova(lm(as.matrix(x)~1, x), idata=ibdat, idesign=~bfct)) mcse.b <- list() mcse.b <- lapply(adatlist, function(x) holm.mc(x, datw=T, psd=F, paired=T)) seffect.b <- lapply(seffect.blist, function(x) Astat(x)$unv) seffect.b # 単純主効果 mcse.b # 多重比較 ## 交互作用対比 ### 処理用オブジェクト icreslist <- list(); n <- 1 lisnm <- vector() cmba <- combn(anms, 2) cmbb <- combn(bnms, 2) ### 交互作用対比の分析 for (i in 1:ncol(cmba)) { x1 <- adatlist[[cmba[1,i]]] x2 <- adatlist[[cmba[2,i]]] for (j in 1:ncol(cmbb)) { y1 <- x1[cmbb[,j]] y2 <- x2[cmbb[,j]] dattmp <- data.frame(y1, y2) names(dattmp) <- c(paste(cmba[1,i], cmbb[,j], sep=""), paste(cmba[2,i], cmbb[,j], sep="")) afctic <- factor(rep(cmba[,i], each=2)) bfctic <- factor(rep(cmbb[,j], 2)) idatic <- data.frame(afctic, bfctic) Anovaresic <- Anova(lm(as.matrix(dattmp)~1, dattmp), idata=idatic, idesign=~afctic*bfctic) Aresic <- Astat(Anovaresic)$unv lisnm <- paste(paste(paste(cmba[1,i], cmbb[,j], sep=""), collapse="_"), "_vs._",paste(paste(cmba[2,i], cmbb[,j], sep=""), collapse="_"), sep="") icreslist[[lisnm]] <- Aresic; n <- n+1 } } #names(icreslist) <- lisnm icreslist tmp <- lapply(icreslist, function(x) x[4,]) tmp2 <- t(data.frame(tmp)) pvholm <- p.adjust(tmp2[,6], "holm") icres <- cbind(tmp2, pvholm) colnames(icres) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") class(icres) <- "anova" icres ## プールされた誤差項で分析してみる。多重比較はやめておこう Ares Aunv ### それぞれの誤差項をとりだしておく (mse.a <- (Aunv[2,3]+Aunv[4,3])/(Aunv[2,4]+Aunv[4,4])) (mse.adf <- (Aunv[2,4]+Aunv[4,4])) (mse.b <- (Aunv[3,3]+Aunv[4,3])/(Aunv[3,4]+Aunv[4,4])) (mse.bdf <- (Aunv[3,4]+Aunv[4,4])) (mse.ab <- (Aunv[4,3])/(Aunv[4,4])) (mse.abdf <- (Aunv[4,4])) ## プールされた誤差項で単純主効果 ## ### 水準別誤差項の結果を利用する # bにおけるAの単純効果 tmp <- list() tmp <- lapply(seffect.a, function(x) Astat(x)$unv[2,]) tmp2 <- t(data.frame(tmp)) pvholm <- p.adjust(1-pf((tmp2[,1]/tmp2[,2])/mse.a, tmp2[,2], mse.adf), "holm") seffect.a.pl <- cbind(tmp2[,1:2], mse.a*mse.adf, mse.adf, mse.a, (tmp2[,1]/tmp2[,2])/mse.a, 1-pf((tmp2[,1]/tmp2[,2])/mse.a, tmp2[,2], mse.adf), pvholm) colnames(seffect.a.pl) <- c("ss", "nmdf", "errorss", "dendf", "mse", "fv", "pv", "Pr(>F).holm") rownames(seffect.a.pl) <- paste("a effect at ", bnms, sep="") class(seffect.a.pl) <- "anova" seffect.a.pl # Bの効果 at a水準 tmp <- list() tmp <- lapply(seffect.b, function(x) Astat(x)$unv[2,]) tmp2 <- t(data.frame(tmp)) pvholm <- p.adjust(1-pf((tmp2[,1]/tmp2[,2])/mse.b, tmp2[,2], mse.bdf), "holm") seffect.b.pl <- cbind(tmp2[,1:2], mse.b*mse.bdf, mse.bdf, mse.b, (tmp2[,1]/tmp2[,2])/mse.b, 1-pf((tmp2[,1]/tmp2[,2])/mse.b, tmp2[,2], mse.bdf), pvholm) colnames(seffect.b.pl) <- c("ss", "nmdf", "errorss", "dendf", "mse", "fv", "pv", "Pr(>F).holm") rownames(seffect.b.pl) <- paste("b effect at ", anms, sep="") class(seffect.b.pl) <- "anova" seffect.b.pl ## プールされた誤差項で交互作用対比…交互作用項の誤差項を使えばいいのか? icres.pltp <- icres[,1:2] pl.mss <- icres[,1]/icres[,2] pl.errorss <- mse.ab*mse.abdf pv.holm <- p.adjust(1-(pf(pl.mss/mse.ab, icres[,2], mse.abdf)), "holm") icres.pl <- cbind(icres.pltp, pl.errorss, mse.abdf, pl.mss/mse.ab, 1-(pf(pl.mss/mse.ab, icres[,2], mse.abdf)), pv.holm) colnames(icres.pl) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") class(icres.pl) <- "anova" icres.pl
PR
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669") source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1273264153") ## データ ## 参考: http://www.psychology.emory.edu/clinical/mcdowell/PSYCH560/mixed.htm 感謝。 dat <- data.frame( cnd = factor(rep(c("wt", "atp", "trt"), each=4)), pre=c(13, 10, 13, 4, 5, 8, 14, 12, 13, 9, 14, 8), hlf=c(14, 11, 19, 12, 10, 15, 16, 21, 24, 22, 22, 18), pst=c(20, 14, 21, 15, 21, 24, 23, 26, 30, 24, 28, 28), fu=c(17, 15, 18, 14, 17, 22, 23, 26, 28, 22, 28, 27) ) by(dat[,2:5], list(dat[,1]), mean) by(dat[,2:5], list(dat[,1]), sd) ## とりあえずプロット x <-aggregate(dat[2:5], list(dat[,1]), mean) gx <- as.matrix(x[2:5]) rownames(gx) <- names(dat[,1]) barplot(gx, beside=T) dev.off() ## 水準名とか ncl <- ncol(dat) anms <- levels(dat[,1]) nanms <- sapply(anms, function(x) sum(x==dat[,1])) cmba <- combn(anms, 2) bnms <- names(dat[2:ncl]) mmbs <- mean(aggregate(dat[,2:ncl], list(dat[,1]), mean)[,2:ncl]) cmbb <- combn(bnms, 2) ## 全体の分散分析 options(contrasts = c("contr.sum", "contr.sum")) lmres <- lm(as.matrix(dat[,2:ncl]) ~ dat[,1] , data = dat) bfact <- factor(bnms) idat <- data.frame(bfact) library(car) Anovares <- Anova(lmres, idata = idat, idesign = ~ bfact) (Ares <- Astat(Anovares)$unv) ## 要因の主効果の多重比較: 参加者間要因 val <- rowMeans(dat[,2:ncl]) a <- dat[,1] factmc.a <- holm.mc(data.frame(a, val)) factmc.a ## 要因の主効果の多重比較: 参加者内要因 factmc.blist <- list() ps <- vector() library(car) for (i in 1:ncol(cmbb)) { mcbfact <- factor(cmbb[,i]) mcidata <- data.frame(mcbfact) mclmres <- lm(as.matrix(dat[,c(cmbb[,i])])~dat[,1], dat) mcAnovares <- Anova(mclmres, idata=mcidata, idesign=~mcbfact) mcAres <- Astat(mcAnovares)$unv mcmse <- mcAres[2,3]/mcAres[2,4] mcdf <- mcAres[2,4] den1 <- (mean(1/nanms)/nlevels(dat[,1]))*2 diff <- mmbs[cmbb[1,i]] - mmbs[cmbb[2,i]] tv <- diff/sqrt(mcmse*den1) ps[i] <- pt(abs(tv), mcdf, lower.tail = F)*2 resv <- c(mmbs[cmbb[1,i]], mmbs[cmbb[2,i]], diff, mcmse, mcdf, tv, pt(abs(tv), mcdf, lower.tail = F)*2) factmc.blist [[i]] <- resv } factmc.b <- t(data.frame(factmc.blist )) ps.holm <- p.adjust(ps, "holm") factmc.b <- cbind(factmc.b, ps.holm) colnames(factmc.b) <- c("m1", "m2", "diff", "mse", "df", "tv", "pv", "Pr(>|t|).holm") rownames(factmc.b) <- paste(cmbb[1,], "_", cmbb[2,], sep="") class(factmc.b) <- "anova" factmc.b ## 単純主効果 ## ## 水準別誤差項で単純主効果 ## ## b (参加者内水準) におけるaの効果とその多重比較 seffect.a <- list() seffect.a <- lapply(dat[,2:ncl], function(x) Anova(lm(x~a, dat))) mcse.a <- list() for (nm in bnms) { val <- dat[nm] x <- dat[,1] mcse.a[[nm]] <- holm.mc(data.frame(x, val)) } names(seffect.a) <- paste(names(dat)[1], " effect at ", bnms, sep="") names(mcse.a) <- paste(names(dat)[1], " effect at ", bnms, sep="") seffect.a # 単純主効果 mcse.a # その多重比較 ## a (参加者間水準) におけるBの効果とその多重比較 seffect.b <- list(); seffect.b <- list() mcse.b <- list() for (nm in anms) { dattmp <- subset(dat, dat[,1]==nm) Anovares.sepa <- Anova(lm(as.matrix(dattmp[,2:ncl])~1, data=dattmp), idata=idat, idesign=~bfact) seffect.b[[nm]] <- Anovares.sepa mcse.b[[nm]] <- holm.mc(dattmp[,2:ncl], datw=T, psd=F, paired=T) } seffect.b <- lapply(seffect.b, function(x) Astat(x)$unv) names(seffect.b) <- paste(names(dat)[2], " effect at ", anms, sep="") names(mcse.b) <- paste(names(dat)[2], " effect at ", anms, sep="") seffect.b # 単純主効果 mcse.b # 多重比較 ## 交互作用対比 ### 処理用オブジェクト iclist <- list() ps <- vector() rnms <- vector() cmba cmbb icn <- 1 ### 交互作用対比の分析 for (i in 1:ncol(cmba)) { dattmpa <- subset(dat, dat[,1]==cmba[1,i]|dat[,1]==cmba[2,i]) for (j in 1:ncol(cmbb)) { dattmp <- data.frame(dattmpa[1], dattmpa[cmbb[,j]]) bfacttmp <- factor(cmbb[,j]) idattmp <- data.frame(bfacttmp) lmrestmp <- lm(as.matrix(dattmp[,2:3])~dattmp[,1], dattmp) Anovarestmp <- Anova(lmrestmp, idata=idattmp, idesign=~bfacttmp) Arestmp <- Astat(Anovarestmp)$unv iclist[[icn]] <- Arestmp[3,]; ps[icn] <- Arestmp[3,6]; rnms[icn] <- paste(paste(cmba[,i], collapse=""), " vs. ", paste(cmbb[,j], collapse=""), sep="") icn <- icn+1 } } icres <- t(data.frame(iclist)) ps.holm <- p.adjust(ps, "holm") icres <- cbind(icres, ps) colnames(icres) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") rownames(icres) <- rnms class(icres) <- "anova" icres # それぞれのmseはicres[,3]/icres[,4] ## プールされた誤差項で分析してみる Ares ### それぞれの誤差項をとりだしておく mse.a <- Ares[1,3]/Ares[1,4] # 参加者間要因のmse: aの主効果の検定に使う df.a <- Ares[1,4] mse.ap <- sum(Ares[c(1,3), 3])/sum(Ares[c(1,3), 4]) # 参加者間要因を含むプールしたmse: aの単純主効果検定につかう df.ap <- sum(Ares[c(1,3), 4]) # mse.aの自由度 mse.b <- Ares[2,3]/Ares[2,4] # 参加者内要因のプールしたmse df.b <- Ares[2,4] # 参加者内要因の自由度 mse.ab <- Ares[3,3]/Ares[3,4] # 交互作用項のプールしたmse: bの単純主効果検定に使う df.ab <- Ares[3,4] # mse.abの自由度 ## 要因の主効果の多重比較: 参加者間要因 val <- rowMeans(dat[,2:ncl]) a <- dat[,1] factmc.a.pl <- holm.mc(data.frame(a, val), mse=mse.a, mse.df=df.a) factmc.a.pl ## 要因の主効果の多重比較: 参加者内要因 factmc.b.pllist <- list() ps <- vector() library(car) for (i in 1:ncol(cmbb)) { mcmse <- mse.ab mcdf <- df.ab den1 <- (mean(1/nanms)/nlevels(dat[,1]))*2 diff <- mmbs[cmbb[1,i]] - mmbs[cmbb[2,i]] tv <- diff/sqrt(mcmse*den1) ps[i] <- pt(abs(tv), mcdf, lower.tail = F)*2 resv <- c(mmbs[cmbb[1,i]], mmbs[cmbb[2,i]], diff, mcmse, mcdf, tv, pt(abs(tv), mcdf, lower.tail = F)*2) factmc.b.pllist [[i]] <- resv } factmc.b.pl <- t(data.frame(factmc.b.pllist )) ps.holm <- p.adjust(ps, "holm") factmc.b.pl <- cbind(factmc.b.pl, ps.holm) colnames(factmc.b.pl) <- c("m1", "m2", "diff", "mse", "df", "tv", "pv", "Pr(>|t|).holm") rownames(factmc.b.pl) <- paste(cmbb[1,], "_", cmbb[2,], sep="") class(factmc.b.pl) <- "anova" factmc.b.pl ## プールされた誤差項で単純主効果 ## ## b (参加者内水準) におけるAの単純効果 seffect.a.pllist <- list() for (nm in bnms) { Anovares.se <- Anova(lm(dat[,nm]~a, dat)) Ares.se <- Astat(Anovares.se)$unv seffect.a.pllist[[nm]] <- c(unlist(Ares.se[1,1:2]), mse.ap, df.ap, ((Ares.se[1,1]/Ares.se[1,2])/mse.ap), 1-pf(((Ares.se[1,1]/Ares.se[1,2])/mse.ap), Ares.se[1,2], df.ap)) } seffect.a.pl <-t(data.frame(seffect.a.pllist)) colnames(seffect.a.pl) <- c("ss", "nmdf", "mse", "dendf", "fv", "Pr(>F)") rownames(seffect.a.pl) <- paste(names(dat)[1], " effect at ", bnms, sep="") class(seffect.a.pl) <- "anova" seffect.a.pl ## 多重比較は勘弁してやろう ## a (参加者間水準) におけるBの単純効果 seffect.b.pllist <- list() for (nm in anms) { dattmp <- subset(dat, dat[,1]==nm) Anovares.se <- Anova(lm(as.matrix(dattmp [,2:ncl])~1, data=dattmp) , idata=idat, idesign=~bfact, type=2) Ares.se1 <- Astat(Anovares.se)$unv if (rownames(Ares.se1)[1]=="(Intercept)") {Ares.se <- Ares.se1[2:nrow(Ares.se1),]} else {Ares.se <- Ares.se1} seffect.b.pllist[[nm]] <- c(unlist(Ares.se[1:2]), mse.b, df.b, ((Ares.se[1]/Ares.se[2])/mse.b), 1-pf(((Ares.se[1]/Ares.se[2])/mse.b), Ares.se[2], df.b)) } seffect.b.pl <-t(data.frame(seffect.b.pllist)) colnames(seffect.b.pl ) <- c("ss", "nmdf", "mse", "dendf", "fv", "Pr(>F)") rownames(seffect.b.pl) <- paste(names(dat)[2], " effect at ", anms, sep="") class(seffect.b.pl ) <- "anova" seffect.b.pl ### プールされた誤差項での交互作用対比 ### 交互作用項のを使えばいいのだろうか? icres.pltp <- icres[,1:2] pl.mss <- icres[,1]/icres[,2] pl.errorss <- mse.ab*df.ab pv.holm <- p.adjust(1-(pf(pl.mss/mse.ab, icres[,2], df.ab)), "holm") icres.pl <- cbind(icres.pltp, pl.errorss, df.ab, pl.mss/mse.ab, 1-(pf(pl.mss/mse.ab, icres[,2], df.ab)), pv.holm) colnames(icres.pl) <- c("ss", "nmdf", "errorss", "dendf", "fv", "pv", "Pr(>F).holm") class(icres.pl) <- "anova" icres.pl
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276622688") source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669") source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1273264153") ## データ ## 参考: http://www.psychology.emory.edu/clinical/mcdowell/PSYCH560/psych560.htm 感謝。 dat <- data.frame( ps = factor(rep(c("ws", "ch", "sk"), each=3, time=3)), cnd = factor(rep(c("nt", "sp", "ms"), each=9)), val=c( c(5,4,5,4,5,3,3,5,4), c(5,6,3,4,6,3,12,10,13), c(10,12,16,11,10,12,13,12,10) )) tapply(dat[,3], list(dat[,1], dat[,2]), mean) tapply(dat[,3], list(dat[,1], dat[,2]), sd) ## 水準名とか anms <- levels(dat[,1]) bnms <- levels(dat[,2]) ## 等分散性の検定 grp <- factor(paste(dat[,1], dat[,2], sep="")) val <- dat[,3] library(car) levene.test(val, grp) ## とりあえずプロット x <- tapply(dat[,3], list(dat[,1], dat[,2]), mean) barplot(x, beside=T) dev.off() ## 全体の分散分析 lmres <- lm(val~ps*cnd, contrasts=list(ps=contr.sum, cnd=contr.sum), data=dat) library(car) Anovares <- Anova(lmres) Anovares errordf <- Anovares[4,2]; errorss <- Anovares[4,1]; mse <- errorss/errordf ## 要因の主効果の多重比較 fact.mc(dat) ## 単純主効果 seffect(dat)[1:2] ## 単純主効果の多重比較 mcse.a <- list() mcse.b <- list() ## b水準のa要因の効果 for (nm in bnms) { x <- subset(dat, dat[2]==nm)[,c(1,3)] mcse.a[[nm]] <- holm.mc(x, mse=mse, errordf) } names(mcse.a) <- paste(names(dat)[1], " effect at ", bnms, sep="") mcse.a ## a水準のb要因の効果 for (nm in anms) { x <- subset(dat, dat[1]==nm)[,c(2,3)] mcse.b[[nm]] <- holm.mc(x, mse=mse, errordf) } names(mcse.b) <- paste(names(dat)[2], " effect at ", anms, sep="") mcse.b ## 交互作用対比 ## データフレームの分割 cmba <- combn(anms,2) cmbb <- combn(bnms,2) n <- 1 nms <- vector() datlist <- list() for (i in 1: ncol(cmba)) { for (j in 1:ncol(cmbb)) { atf <- (dat[,1]==cmba[1,i]|dat[,1]==cmba[2,i]) btf <- (dat[,2]==cmbb[1,j]|dat[,2]==cmbb[2,j]) nms[n] <- paste(paste(cmba[1,i], "-", cmba[2,i], sep=""), " vs. ", paste(cmbb[1,j], "-", cmbb[2,j], sep=""), sep="") datlist[[n]] <- subset(dat, atf&btf); n <- n+1 } } names(datlist) <- nms ### 交互作用対比の分析 iclist <- lapply(datlist, function(x) Anova(lm(val~ps*cnd, x), error=lmres)) tmp <- lapply(iclist, function(x) unlist(x[3,])) tmp2 <- as.matrix(t(sapply(tmp, rbind))) ps <- tmp2[,4] ps.holm <- p.adjust(ps, "holm") icres <- cbind(tmp2[,1:2], errorss, errordf, mse, tmp2[,3:4], sqrt(tmp2[,3]), ps.holm) colnames(icres) <- c("ss", "nmdf", "errorss", "errordf", "mse", "fv", "pv", "tv", "Pr(>F).holm") class(icres) <- "anova" icres # こんな交互作用対比のやり方もありえんわなー
有意水準調整型の多重比較の関数 holm.mc
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669")
## 使い方の覚書
# 与えるデータは以下のように1列目に因子変数、2列目に数値変数。横長のデータはdatw=TRUEと指定する。横長の場合は変数名が因子名で、データは数値のみ
# デフォルトでプールされた標準偏差で多重比較する
# mse=で数値を入れれば、これを元にプールされた標準偏差を使って検定する
# 調整はデフォルトでholm。p.adjust.methodにあるものは選べる
# paired=Tのときは個々にt検定を繰り返すだけ
# psd=F, paired=Fで個々の対応なし検定繰り返し
# デフォルトでWelchの検定。var.equal=Tにすると普通のやつ
dat <- data.frame(a = factor(c(rep("a1",8), rep("a2",8), rep("a3",8), rep("a4",8))), result = c(9,7,8,8,12,11,8,13, 6,5,6,3,6,7,10,9, 10,13,8,13,12,14,14,16, 9,11,13,14,16,12,15,14))
dat # 縦長
dat2 <- data.frame(a1=c(9,7,8,8,12,11,8,13), a2=c(6,5,6,3,6,7,10,9), a3=c(10,13,8,13,12,14,14,16), a4=c(9,11,13,14,16,12,15,14))
dat2 #横長
res <- holm.mc(dat) # デフォルト
print.anova(res)
res1 <- holm.mc(dat, mse=5) # 適当にmseを指定する。内部ではsqrt(mse) を計算する。mseを指定した場合はその自由度をmse.df=で指定しないと警告が出る
res1.2 <- holm.mc(dat, mse=5, mse.df=30)
print.anova(res1.2)
res2 <- holm.mc(dat, psd=F, paired=T)
print.anova(res2)
res3 <- holm.mc(dat, psd=F, paired=F)
print.anova(res3)
res4 <- holm.mc(dat, psd=F, paired=F, var.equal=T)
print.anova(res4)
res5 <- holm.mc(dat2, datw=T, paired=T, psd=F) # 横長、対応あり、プールしない。対応ありのときは対象となる2群でプールするので全体では基本的にプールしない (たぶん)
print.anova(res5)
## うーむ、どうも引数の指定がオシャレじゃないな…
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276531669")
## 使い方の覚書
# 与えるデータは以下のように1列目に因子変数、2列目に数値変数。横長のデータはdatw=TRUEと指定する。横長の場合は変数名が因子名で、データは数値のみ
# デフォルトでプールされた標準偏差で多重比較する
# mse=で数値を入れれば、これを元にプールされた標準偏差を使って検定する
# 調整はデフォルトでholm。p.adjust.methodにあるものは選べる
# paired=Tのときは個々にt検定を繰り返すだけ
# psd=F, paired=Fで個々の対応なし検定繰り返し
# デフォルトでWelchの検定。var.equal=Tにすると普通のやつ
dat <- data.frame(a = factor(c(rep("a1",8), rep("a2",8), rep("a3",8), rep("a4",8))), result = c(9,7,8,8,12,11,8,13, 6,5,6,3,6,7,10,9, 10,13,8,13,12,14,14,16, 9,11,13,14,16,12,15,14))
dat # 縦長
dat2 <- data.frame(a1=c(9,7,8,8,12,11,8,13), a2=c(6,5,6,3,6,7,10,9), a3=c(10,13,8,13,12,14,14,16), a4=c(9,11,13,14,16,12,15,14))
dat2 #横長
res <- holm.mc(dat) # デフォルト
print.anova(res)
res1 <- holm.mc(dat, mse=5) # 適当にmseを指定する。内部ではsqrt(mse) を計算する。mseを指定した場合はその自由度をmse.df=で指定しないと警告が出る
res1.2 <- holm.mc(dat, mse=5, mse.df=30)
print.anova(res1.2)
res2 <- holm.mc(dat, psd=F, paired=T)
print.anova(res2)
res3 <- holm.mc(dat, psd=F, paired=F)
print.anova(res3)
res4 <- holm.mc(dat, psd=F, paired=F, var.equal=T)
print.anova(res4)
res5 <- holm.mc(dat2, datw=T, paired=T, psd=F) # 横長、対応あり、プールしない。対応ありのときは対象となる2群でプールするので全体では基本的にプールしない (たぶん)
print.anova(res5)
## うーむ、どうも引数の指定がオシャレじゃないな…
Holm, Scheffe, TukeyHSD, Dunnet あたりがメジャー。あと、ペリの方法が検定力が高いらしい。
基本的に、群間の独立性を仮定している。すなわち、対応なしの検定である。
ただし、Holm等、有意水準調整型の場合は対応ありでも一応使える
dat <- data.frame(a = factor(c(rep("a1",8), rep("a2",8), rep("a3",8), rep("a4",8))), result = c(9,7,8,8,12,11,8,13, 6,5,6,3,6,7,10,9, 10,13,8,13,12,14,14,16, 9,11,13,14,16,12,15,14))
# 平均と合計
aggregate(dat[2], list(dat[,1]), mean)
aggregate(dat[2], list(dat[,1]), sum)
# lmで分析
lmres <- lm(result~a, dat)
library(car)
Anova(lmres)
## 色々な多重比較
# Holm法、プールされていない標準偏差
## こっちと同じ
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1275792703")
val <- dat$result
idv <- dat$a
cbn <- combn(levels(idv), 2)
tresdat <- tobj(t.test(rnorm(10), rnorm(10), paired=F)); tresdat[1,] <- NA # 結果格納用データフレーム
for (i in 1:ncol(cbn)) {
tres <- t.test(val[which(idv==cbn[1,i])], val[which(idv==cbn[2,i])], paired=F)
tob <- tobj(tres)
tresdat[i,] <- tob
}
rownames(tresdat) <- apply(cbn, 2, function(x) paste(x, collapse=","))
tresdat
## Holm法でp値の調整をする
pholms <- p.adjust(tresdat[,"p.vl"], "holm")
(tresdat <- data.frame(tresdat, pholms))
colnames(tresdat) <- c(colnames(tresdat[-ncol(tresdat)]), "Pr(>|t|).Holm")
print.anova(tresdat) ## 有意の星をつける。文字型の変数は01にされる
# Holm法、プールされた標準偏差
## pairwise.t.test
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1275849015")
pairwise.t.test(dat$result, dat$a, p.adj="holm")
ptest(dat$result, dat$a)
## 青木先生の関数。感謝
source("http://aoki2.si.gunma-u.ac.jp/R/src/Bonferroni.R", encoding="euc-jp")
Bonferroni(dat$result, dat$a, method="Holm")
print.anova(Bonferroni(dat$result, dat$a, method="Holm")$result2)
x <- Bonferroni(dat$result, dat$a, method="Holm")$result2[,2]
round(p.adjust(x, "holm"), 5)
# scheffe
## 青木先生のコードより。感謝
source("http://aoki2.si.gunma-u.ac.jp/R/src/scheffe.R", encoding="euc-jp")
ns <- tapply(dat$result, dat$a, length)
ms <- tapply(dat$result, dat$a, mean)
us <- tapply(dat$result, dat$a, var)
scheffe(ns, ms, us, 1,2)
scheffe(ns, ms, us, 1,3)
scheffe(ns, ms, us, 1,4)
scheffe(ns, ms, us, 2,3)
scheffe(ns, ms, us, 2,4)
scheffe(ns, ms, us, 3,4)
## こういう関数も見つけた。
http://www.biw.kuleuven.be/vakken/statisticsbyr/ANOVAbyRr/multiplecompJIMRC.htm
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276112608")
aovres <- aov(result~a, dat)
summary(aovres)
nis <- tapply(dat$result, dat$a, length); nis
ms <- tapply(dat$result, dat$a, mean); ms
mse.df <- aovres$df.residual; mse.df
mse <- sum(aovres$residuals^2)/mse.df; mse
scheffeCI(ms, nis, mse.df, mse, conf=.95)
## ちょっと改造してF値とp値を出すようにした
scres <- scheffeCI2(ms, nis, mse.df, mse, conf=.95)
print.anova(scres)
# Tukey
library(multcomp)
summary(glht(lmres,linfct=mcp(a="Tukey"))) # これはなんかおかしい
summary(glht(aov(lmres),linfct=mcp(a="Tukey"))) # 教科書と合うのはこっち
TukeyHSD(aov(lmres))
## 小塩先生のspssでの分析例をやってみた。感謝
cond <- factor(rep(1:3, each=7))
result <- c(4,1,3,2,2,4,3,6,8,5,9,8,7,7,4,3,4,6,5,5,5)
aovres <- aov(result~cond)
TukeyHSD(aovres)
library(multcomp)
summary(glht(aovres, linfct=mcp(cond="Tukey")))
## TukeyHSDとglhtで微妙に結果が違うのはなんでだろーなー
## Dunnet 対照群は一番上の水準 ("a1")
summary(glht(aov(lmres),linfct=mcp(a= "Dunnett")))
## Williams なんだかよくわからない。水準間に順序が仮定できるときに使うらしい。
summary(glht(aov(lmres),linfct=mcp(a= "Williams")))
## そのうちやろう
# 2要因以上の分散分析で交互作用が出たときはプールされた分散をつかい統計量を算出する
# glhtの使い方。特にTukey
# 各群のサンプルサイズが違う場合の多重比較。
参考
http://www.ibaraki-kodomo.com/toukei/posthoc.html
http://www.gen-info.osaka-u.ac.jp/testdocs/tomocom/tazyu.html
http://home.hiroshima-u.ac.jp/keiroh/maeda/statsarekore/posthoc.html
基本的に、群間の独立性を仮定している。すなわち、対応なしの検定である。
ただし、Holm等、有意水準調整型の場合は対応ありでも一応使える
dat <- data.frame(a = factor(c(rep("a1",8), rep("a2",8), rep("a3",8), rep("a4",8))), result = c(9,7,8,8,12,11,8,13, 6,5,6,3,6,7,10,9, 10,13,8,13,12,14,14,16, 9,11,13,14,16,12,15,14))
# 平均と合計
aggregate(dat[2], list(dat[,1]), mean)
aggregate(dat[2], list(dat[,1]), sum)
# lmで分析
lmres <- lm(result~a, dat)
library(car)
Anova(lmres)
## 色々な多重比較
# Holm法、プールされていない標準偏差
## こっちと同じ
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1275792703")
val <- dat$result
idv <- dat$a
cbn <- combn(levels(idv), 2)
tresdat <- tobj(t.test(rnorm(10), rnorm(10), paired=F)); tresdat[1,] <- NA # 結果格納用データフレーム
for (i in 1:ncol(cbn)) {
tres <- t.test(val[which(idv==cbn[1,i])], val[which(idv==cbn[2,i])], paired=F)
tob <- tobj(tres)
tresdat[i,] <- tob
}
rownames(tresdat) <- apply(cbn, 2, function(x) paste(x, collapse=","))
tresdat
## Holm法でp値の調整をする
pholms <- p.adjust(tresdat[,"p.vl"], "holm")
(tresdat <- data.frame(tresdat, pholms))
colnames(tresdat) <- c(colnames(tresdat[-ncol(tresdat)]), "Pr(>|t|).Holm")
print.anova(tresdat) ## 有意の星をつける。文字型の変数は01にされる
# Holm法、プールされた標準偏差
## pairwise.t.test
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1275849015")
pairwise.t.test(dat$result, dat$a, p.adj="holm")
ptest(dat$result, dat$a)
## 青木先生の関数。感謝
source("http://aoki2.si.gunma-u.ac.jp/R/src/Bonferroni.R", encoding="euc-jp")
Bonferroni(dat$result, dat$a, method="Holm")
print.anova(Bonferroni(dat$result, dat$a, method="Holm")$result2)
x <- Bonferroni(dat$result, dat$a, method="Holm")$result2[,2]
round(p.adjust(x, "holm"), 5)
# scheffe
## 青木先生のコードより。感謝
source("http://aoki2.si.gunma-u.ac.jp/R/src/scheffe.R", encoding="euc-jp")
ns <- tapply(dat$result, dat$a, length)
ms <- tapply(dat$result, dat$a, mean)
us <- tapply(dat$result, dat$a, var)
scheffe(ns, ms, us, 1,2)
scheffe(ns, ms, us, 1,3)
scheffe(ns, ms, us, 1,4)
scheffe(ns, ms, us, 2,3)
scheffe(ns, ms, us, 2,4)
scheffe(ns, ms, us, 3,4)
## こういう関数も見つけた。
http://www.biw.kuleuven.be/vakken/statisticsbyr/ANOVAbyRr/multiplecompJIMRC.htm
source("https://blog.cnobi.jp/v1/blog/user/89d80905c7038b4121822249e9062fba/1276112608")
aovres <- aov(result~a, dat)
summary(aovres)
nis <- tapply(dat$result, dat$a, length); nis
ms <- tapply(dat$result, dat$a, mean); ms
mse.df <- aovres$df.residual; mse.df
mse <- sum(aovres$residuals^2)/mse.df; mse
scheffeCI(ms, nis, mse.df, mse, conf=.95)
## ちょっと改造してF値とp値を出すようにした
scres <- scheffeCI2(ms, nis, mse.df, mse, conf=.95)
print.anova(scres)
# Tukey
library(multcomp)
summary(glht(lmres,linfct=mcp(a="Tukey"))) # これはなんかおかしい
summary(glht(aov(lmres),linfct=mcp(a="Tukey"))) # 教科書と合うのはこっち
TukeyHSD(aov(lmres))
## 小塩先生のspssでの分析例をやってみた。感謝
cond <- factor(rep(1:3, each=7))
result <- c(4,1,3,2,2,4,3,6,8,5,9,8,7,7,4,3,4,6,5,5,5)
aovres <- aov(result~cond)
TukeyHSD(aovres)
library(multcomp)
summary(glht(aovres, linfct=mcp(cond="Tukey")))
## TukeyHSDとglhtで微妙に結果が違うのはなんでだろーなー
## Dunnet 対照群は一番上の水準 ("a1")
summary(glht(aov(lmres),linfct=mcp(a= "Dunnett")))
## Williams なんだかよくわからない。水準間に順序が仮定できるときに使うらしい。
summary(glht(aov(lmres),linfct=mcp(a= "Williams")))
## そのうちやろう
# 2要因以上の分散分析で交互作用が出たときはプールされた分散をつかい統計量を算出する
# glhtの使い方。特にTukey
# 各群のサンプルサイズが違う場合の多重比較。
参考
http://www.ibaraki-kodomo.com/toukei/posthoc.html
http://www.gen-info.osaka-u.ac.jp/testdocs/tomocom/tazyu.html
http://home.hiroshima-u.ac.jp/keiroh/maeda/statsarekore/posthoc.html