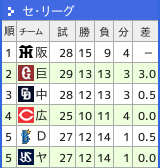
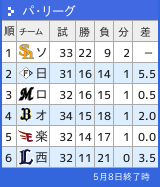
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
Rjpwikiのファイルを読み込むTips参考。感謝
# 複数のデータファイルを一括してリストに読み込む
fnames <- dir(pattern=".csv") # とすると、作業ディレクトリ内のcsvファイルのみを選んでくれる
# fnamesp <- choose.files() # ファイルを選ぶ。パスのベクトルが保存される。リストの要素名には使いづらい
csvlist <- lapply(fnames, read.csv)
names(csvlist) <- fnames
csvlist
# 読み込んだcsvをマージする
n <- length(csvlist)
temp <- csvlist[[1]]
for (i in 2:n) {
temp <- merge(temp, csvlist[[i]], all=T)
}
res <- temp
res
## 青木先生のmerge.allを参考にした。感謝
http://aoki2.si.gunma-u.ac.jp/lecture/mb-arc/arc040/03212.html
merge.all <- function(...)
{
n <- length(list(...))
temp <- list(...)[[1]]
for (i in 2:n) {
temp <- merge(temp, list(...)[[i]], all=TRUE)
}
return(temp)
}
# apply系とchoose.filesを上手く使えば、データ指定から直接マージもできる…かもしれないが、いったんリストにしてRのオブジェクトにしといたほうが個人的にはいいと思う
PR
# MASSパッケージ、mvrnorm関数の覚書
sg <- matrix(c(1, 0.5, 0.5,1), nr=2) # 0.5の相関係数をもつ場合の相関行列
sg
vs <- mvrnorm(n=1000, mu=c(0, 0), Sigma=sg, empirical=T)
x <- vs[,1]
y <- vs[,2]
# プロット
plot(x,y, main="r = .50", pch=20)
abline(lm(y~x))
## よく統計の教科書に載ってる相関の表
# データ生成
library(MASS)
crv <- c(0, 0.2, 0.4, 0.6, 0.8, 1.0)
vrlist <- list()
for (i in 1:6) {
sg <- matrix(c(1, crv[i], crv[i], 1), nr=2)
vs <- mvrnorm(n=300, mu=c(0, 0), Sigma=sg, empirical=T)
vrlist[[i]] <- vs
}
# プロット
par(mfrow=c(2,3))
for (i in 1:6) {
mat <- vrlist[[i]]
x <- mat[,1]
y <- mat[,2]
ttl <- paste("r = ", crv[i], sep="")
plot(x,y, main=ttl, pch=20)
}
# 回帰直線を追加するプロット
win.graph()
par(mfrow=c(2,3))
for (i in 1:6) {
mat <- vrlist[[i]]
x <- mat[,1]
y <- mat[,2]
ttl <- paste("r = ", crv[i], sep="")
plot(x,y, main=ttl, pch=20)
abline(lm(y~x), col="blue", lwd=2)
}
sg <- matrix(c(1, 0.5, 0.5,1), nr=2) # 0.5の相関係数をもつ場合の相関行列
sg
vs <- mvrnorm(n=1000, mu=c(0, 0), Sigma=sg, empirical=T)
x <- vs[,1]
y <- vs[,2]
# プロット
plot(x,y, main="r = .50", pch=20)
abline(lm(y~x))
## よく統計の教科書に載ってる相関の表
# データ生成
library(MASS)
crv <- c(0, 0.2, 0.4, 0.6, 0.8, 1.0)
vrlist <- list()
for (i in 1:6) {
sg <- matrix(c(1, crv[i], crv[i], 1), nr=2)
vs <- mvrnorm(n=300, mu=c(0, 0), Sigma=sg, empirical=T)
vrlist[[i]] <- vs
}
# プロット
par(mfrow=c(2,3))
for (i in 1:6) {
mat <- vrlist[[i]]
x <- mat[,1]
y <- mat[,2]
ttl <- paste("r = ", crv[i], sep="")
plot(x,y, main=ttl, pch=20)
}
# 回帰直線を追加するプロット
win.graph()
par(mfrow=c(2,3))
for (i in 1:6) {
mat <- vrlist[[i]]
x <- mat[,1]
y <- mat[,2]
ttl <- paste("r = ", crv[i], sep="")
plot(x,y, main=ttl, pch=20)
abline(lm(y~x), col="blue", lwd=2)
}
rvlup <- function(x, i, lbl) { # xはラベルとかをくっつけたいベクトル、iはインデックス、lblはラベル。
if(length(i)!=length(lbl))
stop("インデックスとラベルの長さが違います")
lbl2 <- lbl
names(lbl2) <- i
res <- lbl2[x]
names(res) <- NULL
return(res)
}
## 覚書: 条件の変数名をインデックスにする。その上で条件の添え字を元のベクトルで指定する。元のベクトルは当然文字列でないといけない
## stackした後のデータを想定。
tr <- sample(paste("trial", 1:10, sep=""), 20, replace=T)
## それぞれの試行はどんな条件かのインデックス
## サンプルデータ
x1 <- paste("trial", 1:10, sep="")
x2 <- sample(c("high", "low"), 10, replace=T)
## インデックスデータ
(indx <- data.frame(indx=x1, cnd=x2))
## インデックス、条件をそれぞれベクトルにする
indv <- indx[,1]
cndv <- indx[,2]
## 関数rvlupを使う
res <- rvlup(tr, indv, cndv)
res
## 確認
data.frame(tr, res)
indx
## やっぱこっちのほうがわかりやすいかも
trcn <- vector()
for (i in 1:length(tr)) {
x <- tr[i]
xi <- which(indv==x)
res <- as.character(cndv[xi])
trcn[i] <- res
}
data.frame(tr, res)
## match関数を使えばもっと簡単だった...orz
tr
indv
cndv
xi <- match(tr, indv) # trでindvに一致するベクトルの添え字を返す。たとえばtr内の"trial9"はindvの何番目にあるか。これをxiに格納する
cndv[xi] # 条件名ベクトルからxiの添え字に入っているものをとりだす
if(length(i)!=length(lbl))
stop("インデックスとラベルの長さが違います")
lbl2 <- lbl
names(lbl2) <- i
res <- lbl2[x]
names(res) <- NULL
return(res)
}
## 覚書: 条件の変数名をインデックスにする。その上で条件の添え字を元のベクトルで指定する。元のベクトルは当然文字列でないといけない
## stackした後のデータを想定。
tr <- sample(paste("trial", 1:10, sep=""), 20, replace=T)
## それぞれの試行はどんな条件かのインデックス
## サンプルデータ
x1 <- paste("trial", 1:10, sep="")
x2 <- sample(c("high", "low"), 10, replace=T)
## インデックスデータ
(indx <- data.frame(indx=x1, cnd=x2))
## インデックス、条件をそれぞれベクトルにする
indv <- indx[,1]
cndv <- indx[,2]
## 関数rvlupを使う
res <- rvlup(tr, indv, cndv)
res
## 確認
data.frame(tr, res)
indx
## やっぱこっちのほうがわかりやすいかも
trcn <- vector()
for (i in 1:length(tr)) {
x <- tr[i]
xi <- which(indv==x)
res <- as.character(cndv[xi])
trcn[i] <- res
}
data.frame(tr, res)
## match関数を使えばもっと簡単だった...orz
tr
indv
cndv
xi <- match(tr, indv) # trでindvに一致するベクトルの添え字を返す。たとえばtr内の"trial9"はindvの何番目にあるか。これをxiに格納する
cndv[xi] # 条件名ベクトルからxiの添え字に入っているものをとりだす
dat <- data.frame(
a1b1 = rnorm(10),
a1b2 = rnorm(10),
a1b3 = rnorm(10, 100, 10),
a1b4 = rnorm(10, 10, 1),
a2b1 = rnorm(10),
a2b2 = rnorm(10),
a2b3 = rnorm(10, 100, 10),
a2b4 = rnorm(10, 10, 1),
a3b1 = rnorm(10),
a3b2 = rnorm(10),
a3b3 = rnorm(10, 100, 10),
a4b4 = rnorm(10, 10, 1)
)
dat
nlva <- 3 # 自分で入力
nlvb <- 4 # 自分で入力
anms <- c("a1", "a2", "a3") # 自分で入力
bnms <- c("b1", "b2", "b3", "b4") # 自分で入力
ncl <- ncol(dat)
clnm <- 1:ncl
clmt <- matrix(clnm, nlvb)
adatlist <- list()
bdatlist <- list()
for (i in 1:nlva) {
x <- dat[clmt[,i]] ; names(x) <- bnms
adatlist[[i]] <- x
}
names(adatlist) <- anms
for (i in 1:nlvb) {
x <- dat[clmt[i,]] ; names(x) <- anms
bdatlist[[i]] <- x
}
names(bdatlist) <- bnms
adatlist
bdatlist
## サンプルデータ
library(psych)
data(bfi) # psychパッケージ内のデータセット
dat <- bfi
# 各変数ごとに反応の種類を調べる
sapply(dat, function(x) levels(factor(x)))
# 変数ごとに反応や単位が違う場合に使う。データはデータフレームにしておくこと
# library(psych); data(sat.act); sapply(sat.act, function(x) levels(factor(x)))
# 全体の回答数
summary(factor(as.matrix(dat)))
# 一応プロットもしてみる
win.graph()
plot(as.vector(as.matrix(dat)))
win.graph()
hist(as.vector(as.matrix(dat)))
# 各項目ごとの反応数を集計する。
# 有効回答 (NA, Inf, NaN以外の反応) 種類をとりだす
(res <- names(table(as.matrix(dat)))) # table関数はNAとかは集計しないことを利用
resN <- length(res) # 反応の種類数
itemN <- ncol(dat) # 項目数
count.dat <- data.frame(matrix(NA, itemN, resN)) # 集計表用の行列。行が項目、列が反応
rownames(count.dat) <- names(dat)
colnames(count.dat) <- res
for (i in 1:resN){
count.dat[,i] <- sapply(dat, function(x) sum(na.omit(x)==res[i]))
}
count.dat
# NA, NaN, Infも調べる
na <- sapply(dat, function(x) sum(is.na(x))) # NAとNaNがカウントされる
inf <- sapply(dat, function(x) sum(is.infinite(x))) # Infをカウント
# 記述統計とかとくっつける。記述統計はpsychパッケージのdescribe関数で
descriptives <- cbind(data.frame(describe(dat)), count.dat, na, inf)
descriptives
# csvで保存
write.csv(descriptives, file="descriptives.csv", quote=FALSE)